| 天然无序蛋白质:序列 | 您所在的位置:网站首页 › 变性蛋白质的主要特点是什么 › 天然无序蛋白质:序列 |
天然无序蛋白质:序列
|
黄永棋 刘志荣 (北京大学化学与分子工程学院,北京大学理论生物学中心,北京分子科学国家实验室,北京 100871) 天然无序蛋白质:序列-结构-功能的新关系 黄永棋 刘志荣* (北京大学化学与分子工程学院,北京大学理论生物学中心,北京分子科学国家实验室,北京 100871) 天然无序蛋白质是一类新发现的蛋白质,它们在天然条件下没有确定的三维结构,却具有正常的生物学功能,广泛参与信号传递、DNA转录、细胞分裂和蛋白质聚集等重要的生理与病理过程.无序蛋白质的发现是对传统的蛋白质“序列-结构-功能”范式的挑战.在这篇综述里,我们首先回顾了蛋白质的传统范式以及无序蛋白质的发现过程,然后介绍无序蛋白质在结构、序列、功能等方面的特征与相互作用,并以分子识别过程为例,进一步阐述目前国际上对无序蛋白质所具有优势的一些认识与观点.我们还分析了无序蛋白质研究在生命科学和医学等领域的应用前景,并介绍了国内在无序蛋白质领域的研究现状. 天然无序蛋白质;蛋白质折叠;蛋白质相互作用;分子识别;蛋白质结构预测;药物设计 长久以来,蛋白质科学研究基本上遵循的是“序列-结构-功能”的范式,即蛋白质的氨基酸序列决定蛋白质唯一的三维结构,而三维结构则决定了蛋白质的生物学功能.这个范式的思想可以一直追溯到1894年Fischer所提出的锁-钥(lock-and-key)模型[1].Wu[2]与Pauling等[3]也在30年代分别提出过相关的理论.这个范式的关键点是认为蛋白质的三维结构是实现其功能的前提.因此一个蛋白质要发挥其功能,就必须在多肽链被合成出来后快速地(通常处于μs到ms量级)折叠成一个能量较低的有序的三维结构.这样,蛋白质的天然状态(native state)就等同于三维有序结构.在过去的几十年里,蛋白质晶体结构的测定以及蛋白质工程、药物设计、酶学等方面的巨大进展都证实了这个范式的有效性.可以说,结构-功能范式是创造现代蛋白质科学的“大爆炸”[4]. 然而,到了上世纪90年代初,随着实验技术的发展,人们开始发现有些蛋白质在天然状态下并不具有一个确定的三维结构,但依然具有正常的生物学活性.后来,实验上发现的具有这种性质的蛋白质越来越多,并逐渐形成了一类与传统蛋白质范式不同的新的蛋白质类型,称为天然无序蛋白质(以下简称无序蛋白质).英文文献里对这种蛋白质的称呼有多种:intrinsically disordered proteins[5]、natively unfolded proteins[4]、intrinsically unstructured proteins[6],以及natively denatured proteins[7].目前,经实验确认的无序蛋白质的数目已经有不少.Uversky[4]在2002年的综述文章中列出了近百种经过实验验证的、整体上无序或包含有长度超过50个残基的无序片段的无序蛋白质.无序蛋白质数据库DisProt[8]则收录有523种无序蛋白质的详尽信息(2009年1月6日版本).而理论上所预言的无序蛋白质数量则更为巨大.例如,真核生物拥有的无序蛋白质的含量高达27%-41%[9]. 无序蛋白质的发现,与蛋白质研究在几个方面的进展有关[10].第一,基因工程方法的发展使得可以通过基因转录与翻译来制备蛋白质,并通过基因突变、敲除等方法来确定其与功能的关系,这样就避免了无序蛋白质在制备过程中容易被蛋白酶所分解的问题.第二,近年来,很多实验技术(特别是核磁共振(NMR))得到了长足的发展,使得可以在溶液中对蛋白质结构信息进行有效表征,因而对无序蛋白质的检测变得容易.最后,基因组计划的开展产生了大量的序列信息,为通过序列分析来确定蛋白质的有序-无序片段提供了条件. 在这篇综述里,我们将介绍无序蛋白质在结构、序列、功能等方面的特征与相互作用,以及最近几年国内外在这个新兴领域的研究进展. 1 无序蛋白质的结构特点与表征1.1 无序蛋白质的检测方法 天然无序蛋白质的特征是整条链或链的一部分并不具有严格的三维结构,原子位置及主链二面角没有特定的平衡值,而是随着时间发生很大的变化.目前实验上有多种技术可以用来检测无序蛋白质[4,11].X射线晶体衍射图上电子密度的缺失通常是蛋白质晶体中无序片段的标志[4];远紫外圆二色(far-UV circular dichroism,CD)谱可以用来定量估计蛋白质在溶液状态下各种二级结构(如α-螺旋,β-折叠和无规卷曲)的含量,从而可以定性地判断出蛋白质是否处于无序态[4];流体力学方法可以直接测量蛋白质分子的回转半径(radius of gyration,Rg)和扩散速率,而相比于相同分子量的有序蛋白质,无序蛋白质结构相对伸展,回转半径大,扩散较慢,因此可以通过凝胶过滤(gel-filtration)、粘度测定法(viscometry)、小角度X射线散射(small angle X-ray scattering,SAXS)、沉降法(sedimentation)以及动态及静态光学散射的方法来检测无序蛋白质[12];通常无序蛋白质结构柔性大,疏水性氨基酸比较暴露,容易被蛋白酶水解,因此利用蛋白酶水解实验也可以粗略地区分有序蛋白质和无序蛋白质[13-14].最重要的研究无序蛋白质的方法要算是NMR方法了.NMR可以给出氨基酸残基水平上的结构信息.例如,在NMR谱图上,处于无序区域的氨基酸残基所对应的化学位移通常由于分散不好而重叠在一起[15];另外,1H-15N的nuclear overhauser effect(NOE)测量对有序的结构给出正的信号,而对无序的结构则给出负的信号[16].NMR方法本身在这些年来也一直保持着快速的发展,现在已经可以实现对细胞内蛋白质的直接表征[17].综合利用这些实验手段可以将无序蛋白质和有序蛋白质区分开,并可以判断无序蛋白质无序的程度有多大. 1.2 无序蛋白质具有局部的二级结构 从总体上看,无序蛋白质没有一个确定的三维结构,但这并不排除它们可以具有某些局部的二级结构[18-19].Fuxreiter等[20]对24个无序蛋白质复合物进行的统计分析表明,无序蛋白质的有些区域具有很强的形成α-螺旋的倾向,并且这些螺旋的结构与复合物中的结构类似;而有些区域则没有这种强的有序倾向,通常这些区域处于连接(linker)区域.无序蛋白质这些局部的二级结构通常是蛋白质-蛋白质以及蛋白质-DNA相互作用时的结合部位,并且很可能是结合过程的起始位点[21].因此,这些局部的二级结构也被称为molecular recognition motif[22],molecular recognition elements(MoREs)[23],或molecular recognition feature(MoRFs)[24].根据结合部位在无序蛋白质序列上具有较高有序倾向的特点,Oldfield[23]和Mészáros[25]等分别发展了一些方法来预测无序蛋白质与受体结合时的可能结合位点. 1.3 折叠与结合的耦合 尽管在溶液中单独存在时是无序的,很多无序蛋白质在与受体结合时会发生诱导折叠,形成一个更加有序的结构.通常这个过程称为折叠与结合的耦合(coupled folding and binding)[10](图1).其中研究的最多的一个体系是转录因子环腺苷酸应答因子结合蛋白(transcription factor cAMP response-element binding protein,CREB)中的激酶诱导结构域(kinaseinducible domain,KID).KID由60个氨基酸组成,单独存在时只表现出少量的二级结构[19].经过磷酸化之后的KID(pKID)与受体KIX结合,pKID上的Thr119-Pro146区域发生折叠,形成两个α-螺旋[26-27].尽管无序蛋白质与受体结合后变得更加有序,但也并不意味着整个无序蛋白质都发生了折叠,有时候肽链上仍然有一些局部的区域是无序的[28].例如pKID上的C端与N端的序列在结合后就仍然保持无序[26].通过对无序蛋白质在形成复合物状态下的分子内和分子间相互作用进行分析,可以知道无序蛋白质与受体分子之间的相互作用要强于无序蛋白质分子内的相互作用,因此无序蛋白质在结合状态下的有序结构主要是由分子间的相互作用来稳定[29]. 1.4 拥挤环境下的结构 尽管体外实验表明无序蛋白质是一个伸展的无序结构,但由于细胞内存在大量的生物大分子,他们占据了细胞体积的5%-40%,导致细胞的内部空间相当拥挤(crowding)[30].因此,一个很自然的问题是:无序蛋白质在细胞内的拥挤环境下是否仍保持无序?除了直接进行体内实验以外,一个常用的实验策略是用一些可溶性的惰性大分子,如牛血清白蛋白(bovine serum albumin,BSA)、dextran和Ficoll 70等来模拟细胞中的分子环境.例如,无序蛋白质α-突触核蛋白(α-synuclein)在300 g·L-1的BSA存在下的构象与其在稀溶液中的构象基本一样[31].类似地,dextran和Ficoll 70也不能从无序蛋白质p27、c-Fos[32]和dehydrin[33]中诱导出有序结构.相反地,无序蛋白质FlgM的C端在浓度大于400 g·L-1的BSA或卵清蛋白(ovalbumin)条件下变得有序[34].αsynuclein的N端也可以在SDS胶束的表面形成螺旋结构[35].随着NMR方法的发展,体内实验也开始在这种研究中被采用.例如,在E.coli细胞内表达的α-synuclein是无序的[31],而FlgM的C端在E.coli细胞里面会变得有序[34]. 总的来说,已有的实验结果表明分子拥挤对无序蛋白质构象的影响是随体系不同而不同的.分子拥挤可能使无序蛋白质变得有序,也可能没有什么影响,这与具体蛋白质的氨基酸序列有关.从原理上讲,大分子的存在使得溶液中自由空间减小,蛋白质更可能倾向于采取体积较小的构象[36-37].但这种构象可能是有序的折叠态,也可能是一个塌缩的非折叠态.目前对分子拥挤与无序蛋白质构象关系以及其生物学意义的研究还不够深入,需要更多实验和理论分析. 1.5 结构系综
图1 无序蛋白质折叠与结合的耦合示意图Fig.1 Schematic representation of coupled folding-binding process of IDPsThe phosphorylated kinase-inducible domain(pKID)is disordered when free,and forms two helices when binding to the ordered kinase-inducible domain interacting domain(KIX). 无序蛋白质与有序蛋白质的最大不同就是其结构上的柔性.对于有序蛋白质,尽管在特殊情况下(比如和配体结合前后)也可能发生构象变化,但这种变化的势垒通常很高,因此在一般情况下很难发生,只有在配体的作用下,才会诱导出构象的变化(induced-fit).对于无序蛋白质,其结构上的柔性使得构象转变的势垒很低,因此在通常的溶液状态下,无序蛋白质是众多构象共存的体系.描述无序蛋白质的结构需使用系综的概念(图2).目前广泛应用于无序蛋白质结构分析的一种实验方法是残留偶极耦合(residual dipolar couplings,RDC)[38].Shortle等[39]第一次使用RDC方法研究了被尿素变性后的葡萄球菌核酸酶(staphylococcal nuclease)的结构.其后RDC方法在蛋白质无序结构的表征上获得了广泛的应用[40-41].另外,SAXS[42-43]与荧光技术[44-45]也被应用于无序蛋白质结构系综的研究中. 尽管直接的分子动力学模拟可以给出很多关于蛋白质变性态(denatured state)或者无序状态的结构信息[46-49],但由于无序状态的构象空间很大,很多时候普通的模拟方法难以获得完整的结构信息.因此,为了更准确地描述和分析无序蛋白质的结构,一种做法是将实验数据作为约束(restraint)施加到系统上,再进行分子动力学模拟[49-52];另一种获得无序蛋白质结构信息的方法是由Forman-Kay等[53-54]发展的ENSEMBLE程序.该方法可以在NOE,顺磁弛豫增强(paramagnetic relaxation enhancement,PRE),二级化学位移 (secondary chemical shifts),3J耦合(3J-couplings),溶剂可及表面积(solvent-accessible surface areas),或Rg等实验数据的基础上获得无序蛋白质的构象[43,53,55]. 2 无序蛋白质的序列2.1 序列的特点 无序蛋白质在氨基酸序列上具有比较明显的特征.其中最显著的特征是它们的疏水残基含量较低,同时拥有较高的未被中和的电荷,因此不能形成一个稳定的疏水核心[4].在疏水性-电荷图上,无序蛋白质与有序蛋白质明显存在于不同的区域(图3).另外,无序蛋白质氨基酸序列的复杂性也较有序蛋白质的低,序列上常常出现重复的区域[56].不同的氨基酸残基也具有不同的促进无序结构形成的倾向,C, W,Y,I,F,V,L,H,T,N比较有利于有序结构的形成,D,M,K,R,S,Q,P,E有利于无序的形成,而其它残基的作用则比较中性[57].无序蛋白质的出现还与氨基酸残基的预期堆积密度(packing density,用一定距离内的近邻残基数目来表征)有关联[58]:预期堆积密度低的序列倾向于形成无序蛋白质;密度高的倾向于形成淀粉状聚集结构;而预期堆积密度适中的序列则倾向于形成有序球状蛋白. 2.2 无序蛋白质的预测 利用无序蛋白质在序列上所具有的鲜明特征,就可以发展出预测蛋白质是否无序的方法.Romero等[59]在1997年首次对蛋白质无序区域进行预测,他们预测的准确性达到70%.此后,无序蛋白质的预测方法得到了迅速发展.目前应用于无序蛋白质序列预测的方法已经超过50种[60-61],包括PONDR[59], FoldIndex[62],NORSnet[63],DISOPRED[64],GlobPlot[65], DisEMBL[66],IUPed[29],PreLink[67],RONN[68],DRIPPRED[69], SPRITZ[70],POODLE-w[71],Ucon[63],foldUnfold[72]等.这些预测方法的准确性普遍达到85%以上.值得指出的是,目前对无序蛋白质的预测已得到了蛋白质结构预测领域的重视.在著名的蛋白质结构预测比赛Critical Assessment of Structure Prediction(CASP)上,从第五届开始就把对无序蛋白质的预测列为比赛内容之一.在最近的CASP 8上,共有25支队伍参加比赛,对122个目标序列进行预测[73].
图2 无序蛋白质CFTR R-结构域的构象[46]Fig.2 Conformations of the unstructured R-domain of CFTR[46]
图3 有序及无序蛋白质的疏水性-电荷分布[4] Fig.3 Distribution of ordered proteins anddisordered proteins in the mean net charge-mean hydrophobicity space[4]Ordered proteins are represented by filled circles and disordered proteins by open circles.〈H〉:mean hydrophobicity;〈R〉:mean net charge 3 无序蛋白质的生物学功能无序蛋白质在生物体内是普遍存在的[57].例如,利用预测算法DISOPRED2对古细菌、细菌和真核生物的蛋白质组序列进行的分析表明,长度大于30个氨基酸的无序区域在这三个物种中的比例分别为2.0%、4.2%和33.0%[64].这些无序蛋白质承担着怎样的生物学功能呢? 研究无序蛋白质功能的一个常用策略是应用生物信息学的方法,利用2.2节提到的预测算法对目前存在的大量基因库数据进行预测,分析蛋白质的结构无序与蛋白质功能之间的关联.通过这种方法,人们发现无序蛋白质在诸如转录和翻译调控、细胞信号转导、蛋白磷酸化及小分子存储等过程中发挥着重要的作用;另一方面,无序蛋白质又似乎给生物体系带来一些不利的影响,经常与多种疾病联系在一起.Dunker课题组[56]发展了一套预测无序蛋白质的方法PONDR,并研究了无序蛋白质与很多功能的相关性.他们的预测表明在真核生物体内,长度大于30个氨基酸的无序蛋白质片段在转录因子中的含量高达90%以上,在E.coli体内也高达62.5%[74];与信号传导相关的蛋白质中,有66%±6%的蛋白质含有无序片段[75];与人类癌症相关的蛋白质中,有79%±5%的蛋白质无序片段[75];在心血管疾病有关的蛋白质中,无序蛋白质的含量也高达57%± 4%[76].另外,Xie等[77]对Swiss Protein Database中超过二十万个蛋白质进行了系统的分析.他们利用Swiss-Prot里的功能关键字对蛋白质进行功能分类,并用PONDR预测每一类里无序的比例.结果发现在710个功能类别里,有238个类别与无序有较强的正相关(相关度最高的5个是核糖核蛋白、核糖体蛋白、发育蛋白、激素、生长因子),有302个与无序有较强的负相关(相关度最高的是氧化还原酶、转移酶、裂合酶、水解酶、异构酶),而剩下的170个类别与无序没有太多关联.在与疾病有关的17个类别里,有11个与无序有正相关,剩下的6个与有序/无序没什么关联. 另外一个策略是综合利用结构表征手段与生化方法对具体蛋白的性质进行详尽的研究.例如,在细胞分裂的调控方面,两个研究较多的无序蛋白质是p21和p27,它们通过抑制细胞周期蛋白依赖性激酶(cyclin-dependent kinases,Cdks)而起作用[78].又如,研究表明无序结构导致的蛋白质聚集是造成神经退行性疾病(如帕金森氏症和老年痴呆症)的重要原因[79].中国科学院上海生命科学研究院胡红雨课题组[80]在研究α-synuclein聚集过程中发现使蛋白质聚集所必需的核心序列GAV motif(Val66-Val74)位于一个无序的序列区间,如果将包含GAV motif的无序片段嫁接到主体蛋白(host protein)上,就可以使主体蛋白发生聚集.另外,中国医学科学院何维课题组[81]发现胸腺的一种老化相关蛋白质,Rwdd1,在电泳实验中表现出异常慢的迁移率,而且更容易被蛋白酶水解,因而推测Rwdd1很可能是一个无序蛋白质.无序蛋白质还与蛋白质的降解有关.通常蛋白质需要与泛素结合后再由蛋白酶体(proteasome)降解.最近的研究表明有一条降解途径是由蛋白质上C端[82]或者N端[83]的无序区域指导进行的.还有,中国科学技术大学涂晓明课题组[84]利用无序蛋白质预测算法、CD光谱和NMR对原核生物体内的一个泛素类蛋白质Rv2111c的结构进行了广泛分析,证明了Rv2111c是一个新的无序蛋白质. 4 无序蛋白质的优势:以分子识别为例无序蛋白质在真核生物中的含量远高于古细菌与细菌中的含量,表明无序蛋白质在某些方面具有潜在的优势.下面我们以分子识别过程为例,介绍目前国际上对无序蛋白质具有优势的一些认识与观点. 4.1 一个无序蛋白质可以和多个受体结合 无序蛋白质结构上的柔性和在复合物状态下发生无序-有序转变的特性使其能够与其它蛋白质和DNA发生广泛的相互作用.目前,无序蛋白质在分子识别过程中的一个被广泛认同的优势是能够在不同的条件下和多个受体进行专一性结合,即一对多的结合方式.细胞周期蛋白依赖性激酶抑制因子p2lWan/Cipl/sdil对细胞周期调控很重要,CD和NMR表明其N端60个氨基酸序列在游离状态下是无序的,而一旦与受体Cdks结合后,该段序列就形成一个稳定的有序结构[85].p21柔性的无序结构使得其能够和多个激酶复合物cyclin-Cdks结合,如:cyclin A-Cdk2、cyclin E-Cdk2和cyclin D-Cdk4[85].同一段无序蛋白质序列在与不同的受体结合后所形成的结构是可以不同的.例如,转录因子缺氧诱导因子1α (transcription factor hypoxia-inducible factor-1α,HIF1α)的C端有一段无序的区域,当该序列与环腺苷酸应答因子结合蛋白的结合蛋白锌离子结合域(TAZ1)结合后形成一个螺旋结构[86];而与天冬氨酸羟化酶(asparagine hydroxylase)结合后却形成一个高度伸展的结构[87]. 在生物系统里面,各种生理过程和大量的信息传递构成了一个复杂的生物网络.生物网络是一个典型的无尺度网络(scale-free network),其中大多数节点(node)的连接数(连接在节点上的边的个数)都很低;但有少数节点却具有大量的连接,这些节点被称为中心节点(hub nodes)[88].中心节点的多连接特性要求处于这些节点上的蛋白质必须具有某些特殊的性质,以使它们可以和大量的不同蛋白质进行结合.Haynes等[89]应用无序蛋白质的预测方法分别对生物网络中的中心节点蛋白和网络端点蛋白进行了比较,发现在酵母(yeast)、蠕虫(worm)、果蝇(fly)和人类(human)四种真核生物中,中心节点蛋白的无序序列含量均比非中心节点蛋白高.针对中心节点蛋白的深入分析还表明无序蛋白质广泛参与生物分子之间的瞬间结合过程,无序的性质使得结合过程具有专一性和可逆性[90].因此,中心节点蛋白具有大量无序序列的性质是生物网络的一个普遍性质,是中心节点蛋白能够实现大量连接、执行复杂生物功能的一个重要因素[91-92]. 4.2 用较短的序列获得更大的结合界面 由于存在结合与折叠的耦合,人们可以利用NMR和XRD获得无序蛋白质与受体结合后形成的复合物的三维结构,并考察无序蛋白质结合界面的特征.Mészáros等[93]对39个无序蛋白质复合物与72个有序蛋白质复合物进行了比较研究.他们发现,两类复合物在界面大小上没有很显著的区别,但是无序蛋白质的界面占分子表面积的比例更大.另外,无序蛋白质复合物界面间存在更多的疏水-疏水相互作用,因此界面上疏水性氨基酸的比例更高.无序蛋白质平均一个氨基酸与受体蛋白形成更多的界面接触,说明无序蛋白质的柔性使得它们可以在结合的时候避开空间上的限制,更好地适应受体蛋白的表面结构,从而实现最大程度的契合.他们的分析还指出,无序蛋白质复合物的界面通常是由一个连续的氨基酸序列构成的,而有序蛋白质复合物的界面则通常由多个不连续的片段形成[93],这就决定了要形成同样大小的结合界面,无序蛋白质只需要较短的氨基酸序列(图4),从而有利于减小蛋白质分子的体积,减小细胞里的拥挤程度[94]. 4.3 以更快的速率与受体结合
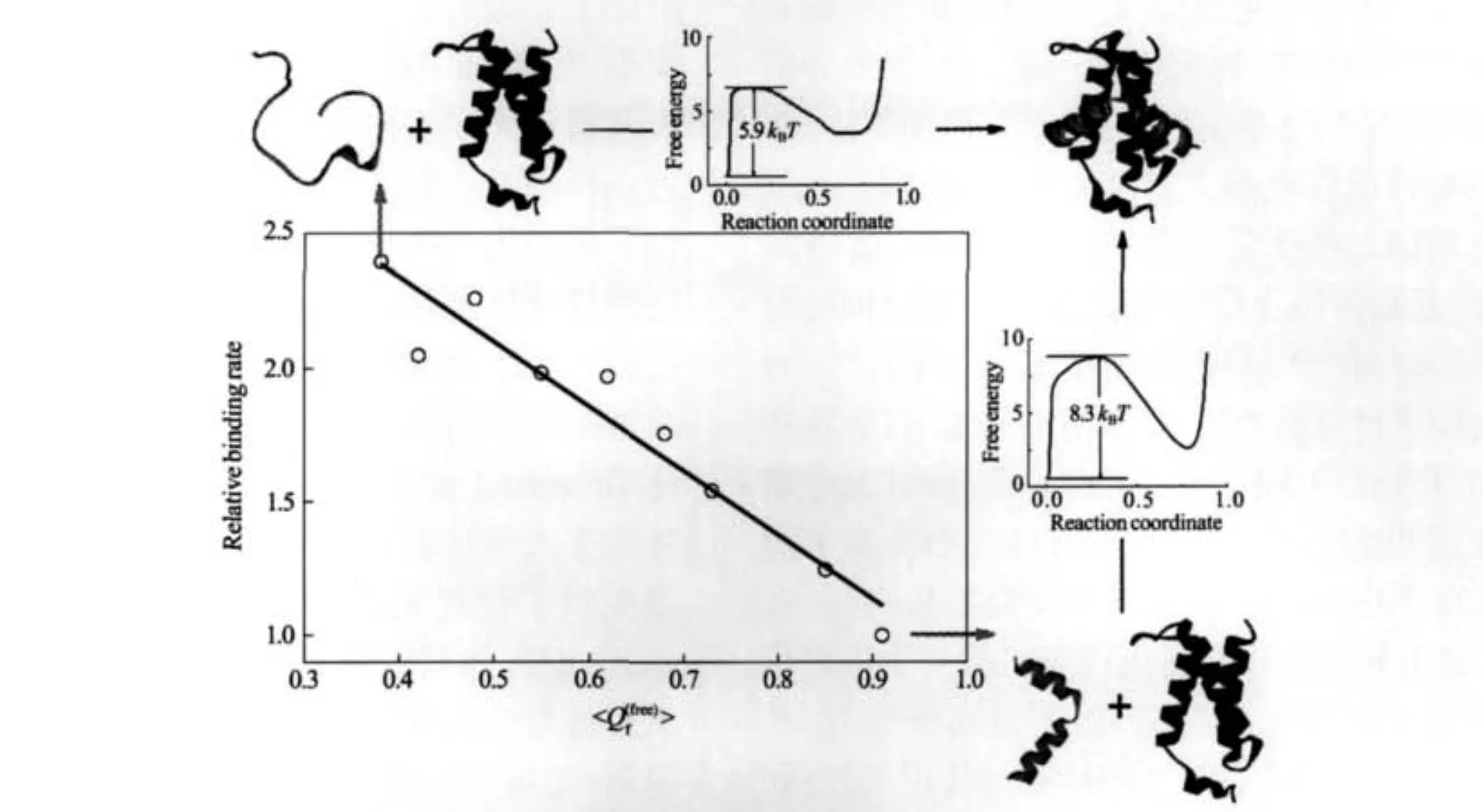
图4 蛋白质二聚体的示意图[94]Fig.4 Schematic representation of protein dimers[94](a)dimers formed by disordered proteins,(b)dimers formed by ordered proteins;The interface areas are the same in(a)and(b). 蛋白质的结合速度在分子识别过程中是至关重要的[95].无序蛋白质的结构柔性对结合速度会有什么样的影响呢?在这方面,一个最被广泛引用的模型是Shoemaker等[96]提出的fly-casting模型.基于无序蛋白质的回转半径比较大的事实,他们提出无序蛋白质在结合过程中具有较大的捕获半径(capture radius),能够从较远的距离与受体发生作用,并启动结合过程,因而具有比有序蛋白质更快的结合速率[96].其它一些理论与模拟研究的结果也支持这个结论.例如,Wang等[97-98]利用解析的方法研究了分子的有序和无序对结合过程的影响,表明无序的体系具有更快的结合速率;Levy等[99]在模拟无序蛋白质与受体DNA结合时指出静电作用使fly-casting效应更加显著;Turjanski等[100]模拟了pKID与 KIX的结合过程,发现增强pKID螺旋的稳定性会使得pKID与KIX的结合速率减慢.但是,对无序蛋白是否真的具有动力学优势,实验上并没有很令人信服的结论,对于fly-casting机制是否适用也存在争议.例如,Crespin等[101]就认为apo-myoglobin的结合动力学不符合fly-casting机制.而且,结合过程中的一些重要因素在fly-casting模型中也没有得到很好的考虑.例如,虽然无序蛋白质的捕获半径比较大,但其扩散速度却会变慢,这个重要因素在flycasting模型中基本是被忽略的.最近,我们对这些问题进行了研究[102].我们比较了45个有序蛋白及35个无序蛋白的结合动力学数据,发现平均而言无序蛋白的结合速度要快2-3倍.进一步地,我们对pKID与KIX的结合过程进行模拟,发现无序蛋白虽然具有较大的捕获半径,但却具有较小的扩散系数,这两个因素基本是互相抵消的,从而导致无序蛋白的捕获速度并不比有序蛋白高;无序蛋白具有较快结合速度的真正原因是其被捕获(形成松散结合状态)后比较容易演化成最终的(紧密)结合结构,而有序蛋白被捕获后却容易逃逸掉,从而需要较多次数的捕获才能最终完成整个的结合过程(图5). 4.4 进行蛋白质修饰和传递信号 转录调节和信号传递过程通常伴随着蛋白质的修饰,如磷酸化、羟基化等.由于结构上的柔性,无序蛋白质更容易把需要修饰的位点暴露到外面,因而有利于修饰的进行[10].Iakoucheva等[103]在对蛋白质磷酸化位点的预测与分析中观察到磷酸化位点周围的氨基酸的性质与无序蛋白质很相近,说明蛋白质磷酸化主要发生在无序的区域内.蛋白质磷酸化可以用来产生超灵敏的结合或者解离信号[104-105],其中研究较多的一个体系,细胞周期蛋白依赖性激酶抑制因子Sic1,就是一个无序蛋白质.KIX与无序蛋白质KID的结合也是通过磷酸化来调节的.KID上的Ser133被磷酸化以后,解离常数Kd从120 μmol· L-1减小到3.1 μmol·L-1,使得KID有效地与KIX结合,从而启动转录过程[106].又如,结构无序性在蛋白质p27调控细胞周期的过程中起着重要的作用.在p27与Cdk2-cyclin A形成的复合物里,p27仍然有部分序列是无序的,这使得被包埋的酪氨酸(Tyr74, Tyr88)能够被激酶磷酸化,然后Cdk2磷酸化p27 C端的苏氨酸(Thr187),从而引发p27的泛素化和降解[78]. 4.5 同时具有高的特异性(specificity)和低的亲和力(affinity)
图5 蛋白质结合过程的热力学与动力学分析[102]Fig.5 Thermodynamic and kinetic analyses of the binding process of protein[102]The average fraction of intramolecular native contacts of pKID in its free form,〈Q(free)〉,was used to quantify the degree of disorder(flexibility)off the model.By scaling the intra-molecular interactions,the pKID domain was continuously tuned from the disordered(coil,small〈Q(free)〉)tof the ordered(two helices,great〈Q(free)〉).Interactions between the pKID and KIX were left unchanged.The disordered system showed lowerf binding free energy barrier and greater binding rate than the ordered counterpart. 无序蛋白质将折叠过程与结合过程耦合起来,通过与受体结合来稳定折叠结构,这种耦合作用使得无序蛋白质的结合过程具有高度的特异性和较低的受体亲和力[9].无序蛋白质构象上的柔性,使得其可以很好适应受体的表面环境,根据受体表面的亲疏水性质、静电性质、几何结构性质,实现最大程度的契合从而达到高度的结合特异性.当无序蛋白质与受体结合并发生有序化转变时,体系的构象熵发生损失,而这个损失将由有序化转变以及结合过程中所发生焓降低来补偿,即焓-熵补偿原理,从而使得体系的亲和力降低[107].在我们对pKID与KIX结合过程的模拟中,我们发现随着pKID螺旋结构的减少,复合物的稳定性确实是降低的[102]. 5 无序蛋白质的研究现状5.1 研究内容与分类 经过近20年时间的发展,无序蛋白质研究已经逐渐成为蛋白质研究领域的一个重要组成部分.目前无序蛋白质的研究可以粗略地分成如下几类: (1)发展无序蛋白质的预测算法并对大量数据进行分析.这基本采用的是生物信息学的思路.无序蛋白质的预测在无序蛋白质研究中占据相对大的比重.从1997年报道的第一个无序蛋白质预测算法起,无序蛋白质预测的进展迅速,目前已经有50多种方法可以从不同的角度去预测一条蛋白质序列的无序程度[60-61].除了方法本身的发展外,无序蛋白质预测算法的研究也有助于深入认识无序蛋白质的动态结构及其内在原因.目前预测方法的研究呈现出功能化和专门化的趋势,例如对结合位点[23,25]和氨基酸修饰位点[103]的预测,这将有助于进一步深入分析无序蛋白质的结构与功能的关系.有了预测算法,人们就可以对目前存在的大量基因库数据进行分析,研究蛋白质的结构无序与物种、蛋白功能、蛋白大小、蛋白在细胞内的分布等性质之间的关联.例如对转录因子的分析[74,108]、对蛋白相互作用的分析[109]、对某一类疾病进行分析[76]等.需要指出的是,目前这些研究虽然可以获得无序与功能的关联,但对无序是如何实现这些功能还缺乏深入的认识.当实验上能够获得无序蛋白质与受体结合的复合物结构时,对这些结构的分析也可以获得无序蛋白质与受体之间相互作用的大量信息:形成界面的氨基酸类型、界面大小、亲疏水性、氨基酸之间接触的情况等[93].同一段无序蛋白质序列还可以和多个受体进行结合[85],通过对这些复合物结构的分析比较可以研究无序蛋白质结合多样性的内在原因.由于NMR和XRD方法测定复合物结构的过程需要很长的时间,因此发展复合物结构的预测方法也是一个很重要的研究方向[110-111]. (2)从实验上对具体体系的结构、功能、机制等进行细致的研究.这基本采用的是物理化学与生物化学的方法与思路.虽然实验上确认的无序蛋白质已经有不少,但相对于无序蛋白质在基因组中所占的比重来说,这些工作还有很大的空间可以发展.实验上主要是发现新的无序蛋白质并研究其功能,例如,对Calmodulin调控因子PEP-19[112]、原核生物的类泛素蛋白Pup[113]、肿瘤抑制因子p53[114]的无序结构都有详细的研究.另外也可以对无序结构的构象性质进行表征,确定这些无序构象的二级结构的含量和类型,以及其多样性是如何影响蛋白质功能的[41-42,44-45,50-52,55].值得一提的是,目前对无序蛋白质构象的研究主要集中在无序蛋白质处于游离态的构象,或者是发生了折叠的结合态的构象.其实,即使在结合态中,仍然有相当的无序蛋白质区域是没有发生折叠的[26,28],目前还没有对这部分区域的构象进行专门研究的报道. (3)理论与模拟研究.与前面两类研究相比,对无序蛋白质的物理、化学性质的计算机模拟及理论研究则相对要少一些.在这方面一个较早的工作是DeLisi等[115]在1994年作出的.他们提出了一个理论框架用于计算配体与蛋白质结合时的自由能变化,并考虑了无序结构带来的柔性与位形熵的问题.另一个比较有影响的工作是前面介绍的fly-casting模型[96].Wang等[97-98]则通过一系列的理论工作详细地分析了无序蛋白质的折叠与结合的耦合过程.最近一项基于基本热力学关系的理论工作在定量上分析了无序蛋白质与功能的关联,并指出无序蛋白质在酶催化以及相互作用很弱的蛋白-蛋白相互作用过程中广泛出现的可能性较小[107].通过简单的格点模型也可以对结合过程进行分析[116],而连续的粗粒化Gō-model更是被广泛用来分析无序蛋白质与受体的热力学与动力学过程,对折叠与结合的耦合过程提供了更深刻的认识[99,100,102,117-118].我们小组对pKID-KIX体系的模拟[102]就属于这类研究.全原子模拟也是非常有用的研究手段,不过受计算能力的限制,对蛋白质结合的研究经常是通过分析复合物的解离过程而进行的[119-120].例如,Chen等[120]对无序蛋白质p53与有序蛋白质MDM2,无序蛋白质PAZ与siRNA[121]和无序蛋白质TISlldTZF与mRNA[122]结合过程中折叠结合的耦合机理进行了大量的研究,模拟的结果与实验结果一致. 5.2 国内无序蛋白质领域的研究现状 无序蛋白质概念在国际上的提出已经引起了国内同行的关注.中国科学院上海生命科学研究院的王克夷[123]在国内期刊《生命的化学》上发表文章介绍了无序蛋白质的概念.中国科学技术大学涂晓明课题组[84]发现原核生物体内的泛素类蛋白质Rv2111c是一个新的无序蛋白质.中国医学科学院何维课题组[81]发现胸腺的一种老化相关蛋白质Rwdd1很可能是一个无序蛋白质.中国科学院上海生命科学研究院胡红雨课题组[80]则发现核心序列GAV motif只有处于无序的序列区间才能使蛋白质发生聚集.哈尔滨工程大学的王科俊课题组与Dunker等人合作,开展了无序蛋白质预测算法的研究并发表了综述文章[61].上海交通大学陈海峰课题组[124]采用全原子分子动力学模拟的方法对KID蛋白的磷酸化、无序蛋白质与RNA的结合过程[121-122]等进行研究,取得了良好效果.德州学院王吉华课题组[125]发展针对无序蛋白的模拟方法,研究无序蛋白质的结构特征、构象变化、热力学和动力学性质,探索其结构与功能,尤其是与疾病的关系[126].香港科技大学的Lee等[127]研究EWS癌蛋白,发现其无序结构的芳香族残基侧链对转移活性起着关键作用.我们课题组[102]主要关注无序蛋白质的构象以及无序蛋白质在结合过程中的热力学和动力学性质,发现无序蛋白质在结合过程中的自由能势垒比有序蛋白质低,结合速率快. 6 无序蛋白质研究的潜在应用对无序蛋白质的研究具有很多潜在的应用: 第一,有助于理解蛋白质的折叠过程.蛋白质的折叠过程是从一个解折叠态出发向折叠态演化的过程,但通常蛋白质的解折叠态是通过化学变性剂或者高温使得蛋白质变性得到的,而处于变性条件下的解折叠构象可能与处于非变性条件下的解折叠构象是不同的.无序蛋白质在天然状态下就是非折叠的,对其构象的研究可以为探索蛋白质的解折叠构象提供重要的信息,从而有助于理解蛋白质的折叠机理,尤其是折叠过程的初始阶段[128]. 第二,帮助确定蛋白质的结构.目前我们拥有的蛋白质序列信息已经超过四百万条,而PDB数据库里记录的蛋白质结构只有约四万个,也就是说大约只有1%的蛋白质的结构被测定了.根据估计,多达70%的蛋白质利用目前的实验方法是无法确定其结构的[129].因此,无序结构预测可以帮助人们更好地排除那些不适合结构测定的蛋白质.另外,无序预测还可以帮助解决结构测定过程中遇到的问题.例如,全长的NEIL1蛋白质在各种条件下都不能结晶.对其进行无序结构预测时发现在C端有一段106个氨基酸的无序片段,在将C端的100个氨基酸去掉后便顺利获得了NEIL1的结晶[130]. 第三,有助于蛋白质设计.蛋白质设计的主要目标是产生新的蛋白质结构类型与功能,而目前的设计工作主要是基于经验和已有的蛋白质结构[131-132].蛋白质结构预测是蛋白质设计的一个重要方面,但预测过程的计算量通常较大[133],并且在缺乏同源结构和相关的实验数据的情况下,预测的准确度往往较低.相反,蛋白质无序结构的预测方法多,计算量小,预测的准确度高.因此可以在蛋白质设计的初始阶段对设计的蛋白质序列进行无序结构的预测,筛选出具有高有序倾向的序列,再进行后继的设计过程.另外,通过嫁接活性位点的方式可以使蛋白质产生新功能[134],这种方案也许可以进一步推广,在现有的有序蛋白质中引进无序的功能序列,从而在保持拓扑结构的同时产生新的功能. 第四,无序蛋白质可能成为新的药物靶标.目前药物设计的靶标主要是有序的蛋白质[135].无序蛋白质在生物体广泛存在,其与受体结合的亲和力较低,因此这种相互作用更容易被小分子阻断,从而有可能成为药物设计的靶标.目前研究比较多的体系有p53-MDM2[136-139]和KID-KIX[140].尽管以无序蛋白质为基础的药物设计只是刚刚开始,但这一领域的开拓可能会给药物开发带来重要影响[141]. 7 总结与展望与传统的蛋白质具有确定三维结构的观点不同,无序蛋白质并没有确定的三维结构,但可以具有局部的二级结构.无序蛋白质具有独特的氨基酸组成特点,这些独特的氨基酸序列决定了其无序的结构.结构上的柔性使得无序蛋白质能够更好地进行分子识别,因此无序蛋白质广泛参与了细胞中很多的生理和病理过程,比如信号传递、DNA的转录的调控、细胞分裂的调控和蛋白质聚集引起的疾病过程.无序蛋白质的研究将促进人们重新认识蛋白质的结构和功能关系,也为蛋白质的全新设计和疾病治疗提供新的思路. 无序蛋白质在各种生物体内广泛存在,并且生命形式越高级无序蛋白质的含量越多,这无疑是生物进化的结果,是蛋白质平行进化的产物.有序蛋白质为生物体提供了结构上的支架(scaffold),使得各种酶促反应和生物过程具有高度的选择性;而无序蛋白质或者无序的区域则大大丰富了有序蛋白质的结构和功能,使得有序蛋白质在结构上具有可调节性和功能的多样性[142].两者的紧密结合使得各种错综复杂的生命过程得以有序进行. 经过十几年的发展,人们对无序蛋白质的认识已经有了很大的进步.除了基础研究的深入外,对无序蛋白质的研究还深化了对蛋白质结构稳定性[143]、酶活性[144]等的认识,也为药物靶点的设计提供了新的选择[141].但要真正清楚认识无序蛋白质的序列-结构-功能关系,还有大量的工作要开展. 1 Fischer,E.Ber.Dt.Chem.Ges.,1894,27:2985 2 Wu,H.Chin.J.Physiol.,1931,1:219 3 Mirsky,A.E.;Pauling,L.Proc.Natl.Acad.Sci.U.S.A.,1936, 22:439 4 Uversky,V.N.Protein Sci.,2002,11:739 5 Dunker,A.K.;Brown,C.J.;Lawson,J.D.;Iakoucheva,L.M.; Obradovic′,Z.Biochemistry,2002,41:6573 6 Wright,P.E.;Dyson,H.J.J.Mol.Biol.,1999,293:321 7 Schweers,O.;Schönbrunn-Hanebeck,E.;Marx,A.;Mandelkow, E.J.Biol.Chem.,1994,269:24290 8 Sickmeier,M.;Hamilton,J.A.;LeGall,T.;Vacic,V.;Cortese,M. S.;Tantos,A.;Szabo,B.;Tompa,P.;Chen,J.;Uversky,V.N.; Obradovic,Z.;Dunker,A.K.Nucleic Acids Res.,2007,35:D786 9 Dunker,A.K.;Lawson,J.D.;Brown,C.J.;Williams,R.M.; Romero,P.;Oh,J.S.;Oldfield,C.J.;Campen,A.M.;Ratliff,C. M.;Hipps,K.W.;Ausio,J.;Nissen,M.S.;Reeves,R.;Kang,C.; Kissinger,C.R.;Bailey,R.W.;Griswold,M.D.;Chiu,W.; Garner,E.C.;Obradovic,Z.J.Mol.Graph.Model.,2001,19:26 10 Dyson,H.J.;Wright,P.E.Nat.Rev.Mol.Cell Biol.,2005,6:197 11 Eliezer,D.Curr.Opin.Struct.Biol.,2009,19:23 12 Receveur-Bréchot,V.;Bourhis,J.M.;Uversky,V.N.;Canard,B.; Longhi,S.Proteins,2006,62:24 13 Fontana,A.;Zambonin,M.;de Laureto,P.P.;DeFilippis,V.; Clementi,A.;Scaramella,E.J.Mol.Biol.,1997,266:223 14 Iakoucheva,L.M.;Kimzey,A.L.;Masselon,C.D.;Bruce,J.E.; Garner,E.C.;Brown,C.J.;Dunker,A.K.;Smith,R.D.; Ackerman,E.J.Protein Sci.,2001,10:560 15 Dyson,H.J.;Wright,P.E.Chem.Rev.,2004,104:3607 16 Muchmore,S.W.;Sattler,M.;Liang,H.;Meadows,R.P.;Harlan, J.E.;Yoon,H.S.;Nettesheim,D.;Chang,B.S.;Thompson,C. B.;Wong,S.L.;Ng,S.C.;Fesik,S.W.Nature,1996,381:335 17 Li,C.G.;Charlton,L.M.;Lakkavaram,A.;Seagle,C.;Wang,G. F.;Young,G.B.;Macdonald,J.M.;Pielak,G.J.J.Am.Chem. Soc.,2008,130:6310 18 Yoon,M.K.;Venkatachalam,V.;Huang,A.;Choi,B.S.;Stultz, C.M.;Chou,J.J.Protein Sci.,2009,18:337 19 Radhakrishnan,I.;Pérez-Alvarado,G.C.;Dyson,H.J.;Wright,P. E.FEBS Lett.,1998,430:317 20 Fuxreiter,M.;Simon,I.;Friedrich,P.;Tompa,P.J.Mol.Biol., 2004,338:1015 21 Csizmók,V.;Bokor,M.;Bánki,P.;Klement,T.;Medzihradszky, K.F.;Friedrich,P.;Tompa,K.A.;Tompa,P.Biochemistry,2005, 44:3955 22 Fuxreiter,M.;Tompa,P.;Simon,I.Bioinformatics,2007,23: 950 23 Oldfield,C.J.;Cheng,Y.G.;Cortese,M.S.;Romero,P.; Uversky,V.N.;Dunker,A.K.Biochemistry,2005,44:12454 24 Mohan,A.;Oldfield,C.J.;Radivojac,P.;Vacic,V.;Cortese,M. S.;Dunker,A.K.;Uversky,V.N.J.Mol.Biol.,2006,362:1043 25 Mészáros,B.;Simon,I.;Dosztányi,Z.PLoS Comput.Biol.,2009, 5:e1000376 26 Radhakrishnan,I.;Pérez-Alvarado,G.C.;Parker,D.;Dyson,H. J.;Montminy,M.R.;Wright,P.E.Cell,1997,91:741 27 Sugase,K.;Dyson,H.J.;Wright,P.E.Nature,2007,447:1021 28 Tompa,P.;Fuxreiter,M.Trends Biochem.Sci.,2008,33:2 29 Dosztányi,Z.;Csizómk,V.;Tompa,P.;Simon,I.J.Mol.Biol., 2005,347:827 30 Ellis,R.J.;Minton,A.P.Nature,2003,425:27 31 McNulty,B.C.;Young,G.B.;Pielak,G.J.J.Mol.Biol.,2006, 355:893 32 Flaugh,S.L.;Lumb,K.J.Biomacromolecules,2001,2:538 33 Mouillon,J.M.;Eriksson,S.K.;Harryson,P.Plant Physiol., 2008,148:1925 34 Dedmon,M.M.;Patel,C.N.;Young,G.B.;Pielak,G.J.Proc. Natl.Acad.Sci.U.S.A.,2002,99:12681 35 Croke,R.L.;Sallum,C.O.;Watson,E.;Watt,E.D.; Alexandrescu,A.T.Protein Sci.,2008,17:1434 36 Wang,W.;Xu,W.X.;Levy,Y.;Trizac,E.;Wolynes,P.G.Proc. Natl.Acad.Sci.U.S.A.,2009,106:5517 37 Zhou,H.X.Arch.Biochem.Biophys.,2008,469:76 38 Meier,S.;Blackledge,M.;Grzesiek,S.J.Chem.Phys.,2008, 128:052204 39 Shortle,D.;Ackerman,M.S.Science,2001,293:487 40 Blackledge,M.Prog.Nucl.Magn.Reson.Spectrosc.,2005,46: 23 41 Jensen,M.R.;Markwick,P.R.L.;Meier,S.;Griesinger,C.; Zweckstetter,M.;Grzesiek,S.;Bernadó,P.;Blackledge,M. Structure,2009,17:1169 42 Bernadó,P.;Mylonas,E.;Petoukhov,M.V.;Blackledge,M.; Svergun,D.I.J.Am.Chem.Soc.,2007,129:5656 43 Mittag,T.;Forman-Kay,J.D.Curr.Opin.Struct.Biol.,2007, 17:3 44 Chen,H.M.;Rhoades,E.Curr.Opin.Struct.Biol.,2008,18:516 45 Huang,F.;Rajagopalan,S.;Settanni,G.;Marsh,R.J.; Armoogum,D.A.;Nicolaou,N.;Bain,A.J.;Lerner,E.;Haas,E.; Ying,L.;Fersht,A.R.Proc.Natl.Acad.Sci.U.S.A.,2009,106: 20758 46 Hegedüs,T.;Serohijos,A.W.R.;Dokholyan,N.V.;He,L.H.; Riordan,J.R.J.Mol.Biol.,2008,378:1052 47 Solt,I.;Magyar,C.;Simon,I.;Tompa,P.;Fuxreiter,M.Proteins, 2006,64:749 48 Ganguly,D.;Chen,J.H.J.Am.Chem.Soc.,2009,131:5214 49 Smith,L.J.Methods,2004,34:144 50 Huang,J.R.;Grzesiek,S.J.Am.Chem.Soc.,2010,132:694 51 Lindorff-Larsen,K.;Kristjansdottir,S.;Teilum,K.;Fieber,W.; Dobson,C.M.;Poulsen,F.M.;Vendruscolo,M.J.Am.Chem. Soc.,2004,126:3291 52 Mylonas,E.;Hascher,A.;Bernadó,P.;Blackledge,M.; Mandelkow,E.;Svergun,D.I.Biochemistry,2008,47:10345 53 Choy,W.Y.;Forman-Kay,J.D.J.Mol.Biol.,2001,308:1011 54 Marsh,J.A.;Neale,C.;Jack,F.E.;Choy,W.Y.;Lee,A.Y.; Crowhurst,K.A.;Forman-Kay,J.D.J.Mol.Biol.,2007,367: 1494 55 Marsh,J.A.;Forman-Kay,J.D.J.Mol.Biol.,2009,391:359 56 Romero,P.;Obradovic,Z.;Li,X.H.;Garner,E.C.;Brown,C.J.; Dunker,A.K.Proteins,2001,42:38 57 Radivojac,P.;Iakoucheva,L.M.;Oldfield,C.J.;Obradovic,Z.; Uversky,V.N.;Dunker,A.K.Biophys.J.,2007,92:1439 58 Galzitskaya,O.V.;Garbuzynskiy,S.O.;Lobanov,M.Y.J.Phys.-Condes.Matter,2007,19:285225 59 Romero,P.;Obradovic,Z.;Kissinger,C.;Villafranca,J.E.; Dunker,A.K.Proc.IEEE Int.Conf.Neural Networks,1997,1: 90 60 Ferron,F.;Longhi,S.;Canard,B.;Karlin,D.Proteins,2006,65:1 61 He,B.;Wang,K.J.;Liu,Y.L.;Xue,B.;Uversky,V.N.;Dunker, A.K.Cell Res.,2009,19:929 62 Prilusky,J.;Felder,C.E.;Zeev-Ben-Mordehai,T.;Rydberg,E. H.;Man,O.;Beckmann,J.S.;Silman,I.;Sussman,J.L. Bioinformatics,2005,21:3435 63 Schlessinger,A.;Punta,M.;Rost,B.Bioinformatics,2007,23: 2376 64 Ward,J.J.;Sodhi,J.S.;McGuffin,L.J.;Buxton,B.F.;Jones,D. T.J.Mol.Biol.,2004,337:635 65 Linding,R.;Russell,R.B.;Neduva,V.;Gibson,T.J.Nucleic Acids Res.,2003,31:3701 66 Linding,R.;Jensen,L.J.;Diella,F.;Bork,P.;Gibson,T.J.; Russell,R.B.Structure,2003,11:1453 67 Coeytaux,K.;Poupon,A.Bioinformatics,2005,21:1891 68 Yang,Z.R.;Thomson,R.;McNeil,P.;Esnouf,R.M. Bioinformatics,2005,21:3369 69 Bryson,K.;McGuffin,L.J.;Marsden,R.L.;Ward,J.J.;Sodhi,J. S.;Jones,D.T.Nucleic Acids Res.,2005,33:W36 70 Vullo,A.;Bortolami,O.;Pollastri,G.;Tosatto,S.C.E.Nucleic Acids Res.,2006,34:W164 71 Shimizu,K.;Muraoka,Y.;Hirose,S.;Tomii,K.;Noguchi,T. BMC Bioinformatics,2007,8:78 72 Galzitskaya,O.V.;Garbuzynskiy,S.O.;Lobanov,M.Y. Bioinformatics,2006,22:2948 73 Noivirt-Brik,O.;Prilusky,J.;Sussman,J.L.Proteins,2009,77: 210 74 Liu,J.G.;Perumal,N.B.;Oldfield,C.J.;Su,E.W.;Uversky,V. N.;Dunker,A.K.Biochemistry,2006,45:6873 75 Iakoucheva,L.M.;Brown,C.J.;Lawson,J.D.;Obradovic′,Z.; Dunker,A.K.J.Mol.Biol.,2002,323:573 76 Cheng,Y.G.;LeGall,T.;Oldfield,C.J.;Dunker,A.K.;Uversky, V.N.Biochemistry,2006,45:10448 77 Xie,H.B.;Vucetic,S.;Iakoucheva,L.M.;Oldfield,C.J.; Dunker,A.K.;Uversky,V.N.;Obradovic,Z.J.Proteome Res., 2007,6:1882 78 Galea,C.A.;Wang,Y.;Sivakolundu,S.G.;Kriwacki,R.W. Biochemistry,2008,47:7598a 79 Uversky,V.N.Frontiers Biosci.,2009,14:5188 80 Ji,L.N.;Du,H.N.;Zhang,F.;Li,H.T.;Luo,X.Y.;Hu,J.;Hu, H.Y.Protein J.,2005,24:209 81 Kang,N.;Chen,D.;Wang,L.;Duan,L.;Liu,S.R.;Tang,L.;Liu, Q.F.;Cui,L.X.;He,W.Cell.Mol.Immunol.,2008,5:333 82 Zhang,M.S.;Pickart,C.M.;Coffino,P.EMBO J.,2003,22: 1488 83 Peña,M.M.O.;Melo,S.P.;Xing,Y.Y.;White,K.;Barbour,K. W.;Berger,F.G.J.Biol.Chem.,2009,284:31597 84 Liao,S.H.;Shang,Q.;Zhang,X.C.;Zhang,J.H.;Xu,C.;Tu,X. M.Biochem.J.,2009,422:207 85 Kriwacki,R.W.;Hengst,L.;Tennant,L.;Reed,S.I.;Wright,P. E.Proc.Natl.Acad.Sci.U.S.A.,1996,93:11504 86 Dames,S.A.;Martinez-Yamout,M.;De Guzman,R.N.;Dyson, H.J.;Wright,P.E.Proc.Natl.Acad.Sci.U.S.A.,2002,99: 5271 87 Elkins,J.M.;Hewitson,K.S.;McNeill,L.A.;Seibel,J.F.; Schlemminger,I.;Pugh,C.W.;Ratcliffe,P.J.;Schofield,C.J. J.Biol.Chem.,2003,278:1802 88 Dunker,A.K.;Cortese,M.S.;Romero,P.;Iakoucheva,L.M.; Uversky,V.N.FEBS J.,2005,272:5129 89 Haynes,C.;Oldfield,C.J.;Ji,F.;Klitgord,N.;Cusick,M.E.; Radivojac,P.;Uversky,V.N.;Vidal,M.;Iakoucheva,L.M.PLoS Comput.Biol.,2006,2:e100 90 Singh,G.P.;Ganapathi,M.;Dash,D.Proteins,2007,66:761 91 Patil,A.;Nakamura,H.FEBS Lett.,2006,580:2041 92 Tompa,P.;Szász,C.;Buday,L.Trends Biochem.Sci.,2005,30: 484 93 Mészáros,B.;Tompa,P.;Simon,I.;Dosztányi,Z.J.Mol.Biol., 2007,372:549 94 Gunasekaran,K.;Tsai,C.J.;Kumar,S.;Zanuy,D.;Nussinov,R. Trends Biochem.Sci.,2003,28:81 95 Schreiber,G.;Haran,G.;Zhou,H.X.Chem.Rev.,2009,109: 839 96 Shoemaker,B.A.;Portman,J.J.;Wolynes,P.G.Proc.Natl. Acad.Sci.U.S.A.,2000,97:8868 97 Wang,J.;Zhang,K.;Lu,H.Y.;Wang,E.K.Phys.Rev.Lett., 2006,96:168101 98 Wang,J.;Zhang,K.;Lu,H.Y.;Wang,E.K.Biophys.J.,2006, 91:866 99 Levy,Y.;Onuchic,J.N.;Wolynes,P.G.J.Am.Chem.Soc., 2007,129:738 100 Turjanski,A.G.;Gutkind,J.S.;Best,R.B.;Hummer,G.PLoS Comput.Biol.,2008,4:e1000060 101 Crespin,M.O.;Boys,B.L.;Konermann,L.FEBS Lett.,2005, 579:271 102 Huang,Y.Q.;Liu,Z.R.J.Mol.Biol.,2009,393:1143 103 Iakoucheva,L.M.;Radivojac,P.;Brown,C.J.;O′Connor,T.R.; Sikes,J.G.;Obradovic,Z.;Dunker,A.K.Nucleic Acids Res., 2004,32:1037 104 Ferrell Jr.,J.E.Trends Biochem.Sci.,1996,21:460 105 Borg,M.;Mittag,T.;Pawson,T.;Tyers,M.;Forman-Kay,J.D.; Chan,H.S.Proc.Natl.Acad.Sci.U.S.A.,2007,104:9650 106 Zor,T.;Mayr,B.M.;Dyson,H.J.;Montminy,M.R.;Wright,P. E.J.Biol.Chem.,2002,277:42241 107 Liu,J.T.;Faeder,J.R.;Camacho,C.J.Proc.Natl.Acad.Sci.U. S.A.,2009,106:19819 108 Singh,G.P.;Dash,D.Proteins,2007,68:602 109 Shimizu,K.;Toh,H.J.Mol.Biol.,2009,392:1253 110 Staneva,I.;Wallin,S.J.Mol.Biol.,2009,393:1118 111 Hu,X.;Lee,M.S.;Wallqvist,A.Biochemistry,2009,48:11158 112 Kleerekoper,Q.K.;Putkey,J.A.J.Biol.Chem.,2009,284:7455 113 Chen,X.;Solomon,W.C.;Kang,Y.;Cerda-Maira,F.;Darwin,K. H.;Walters,K.J.J.Mol.Biol.,2009,392:208 114 Wells,M.;Tidow,H.;Rutherford,T.J.;Markwick,P.;Jensen,M. R.;Mylonas,E.;Svergun,D.I.;Blackledge,M.;Fersht,A.R. Proc.Natl.Acad.Sci.U.S.A.,2008,105:5762 115 Vajda,S.;Weng,Z.P.;Rosenfeld,R.;DeLisi,C.Biochemistry, 1994,33:13977 116 Gupta,N.;Irbäck,A.J.Chem.Phys.,2004,120:3983 117 Wang,J.;Lu,Q.;Lu,H.P.PLoS Comput.Biol.,2006,2:e78 118 Levy,Y.;Wolynes,P.G.;Onuchic,J.N.Proc.Natl.Acad.Sci.U. S.A.,2004,101:511 119 Verkhivker,G.M.;Bouzida,D.;Gehlhaar,D.K.;Rejto,P.A.; Freer,S.T.;Rose,P.W.Proc.Natl.Acad.Sci.U.S.A.,2003, 100:5148 120 Chen,H.F.;Luo,R.J.Am.Chem.Soc.,2007,129:2930 121 Chen,H.F.J.Chem.Theory Comput.,2008,4:1360 122 Qin,F.;Chen,Y.;Li,Y.X.;Chen,H.F.J.Chem.Phys.,2009, 131:115103 123 Wang,K.Y.Chem.Life,2006,26:199 [王克夷.生命的化学, 2006,26:199] 124 Chen,H.F.PLoS ONE,2009,4:e6516 125 Cao,Z.X.;Wang,J.H.J.Biomol.Struct.Dyn.,2010,5:651 126 Wang,J.H.;Cao,Z.X.;Li,S.Q.Curr.Comput.Aided Drug Des., 2009,5:280 127 Ng,K.P.;Potikyan,G.;Savene,R.O.V.;Denny,C.T.;Uversky, V.N.;Lee,K.A.W.Proc.Natl.Acad.Sci.U.S.A.,2007,104: 479 128 Smith,L.J.;Fiebig,K.M.;Schwalbe,H.;Dobson,C.M.Folding Des.,1996,1:R95 129 Adams,M.;Joachimiak,A.;Kim,R.;Montelione,G.T.;Norvell, J.J.Struct.Funct.Genomics,2004,5:1 130 Bandaru,V.;Cooper,W.;Wallace,S.S.;Doublie,S.Acta Crystallogr.Sect.D-Biol.Crystallogr.,2004,60:1142 131 Lippow,S.M.;Tidor,B.Curr.Opin.Biotech.,2007,18:305 132 Floudas,C.A.;Fung,H.K.;McAllister,S.R.;Mönigmann,M.; Rajgaria,R.Chem.Eng.Sci.,2006,61:966 133 Kim,D.E.;Blum,B.;Bradley,P.;Baker,D.J.Mol.Biol.,2009, 393:249 134 Liu,S.;Liu,S.Y.;Zhu,X.L.;Liang,H.H.;Cao,A.N.;Chang,Z. J.;Lai,L.H.Proc.Natl.Acad.Sci.U.S.A.,2007,104:5330 135 Zheng,C.J.;Han,L.Y.;Yap,C.W.;Ji,Z.L.;Cao,Z.W.;Chen, Y.Z.Pharmacol.Rev.,2006,58:259 136 Chène,P.Mol.Cancer Res.,2004,2:20 137 Wasylyk,C.;Salvi,R.;Argentini,M.;Dureuil,C.;Delumeau,I.; Abecassis,J.;Debussche,L.;Wasylyk,B.Oncogene,1999,18: 1921 138 Chène,P.;Fuchs,J.;Bohn,J.;García-Echeverría,C.;Furet,P.; Fabbro,D.J.Mol.Biol.,2000,299:245 139 Vassilev,L.T.Cell Cycle,2004,3:419 140 Best,J.L.;Amezcua,C.A.;Mayr,B.;Flechner,L.;Murawsky,C. M.;Emerson,B.;Zor,T.;Gardner,K.H.;Montminy,M.Proc. Natl.Acad.Sci.U.S.A.,2004,101:17622 141 Cheng,Y.;LeGall,T.;Oldfield,C.J.;Mueller,J.P.;Van,Y.Y. J.;Romero,P.;Cortese,M.S.;Uversky,V.N.;Dunker,A.K. Trends Biotech.,2006,24:435 142 Cortese,M.S.;Uversky,V.N.;Dunker,A.K.Prog.Biophys. Mol.Biol.,2008,98:85 143 Horn,J.R.;Kraybill,B.;Petro,E.J.;Coales,S.J.;Morrow,J.A.; Hamuro,Y.;Kossiakoff,A.A.Biochemistry,2006,45:8488 144 Vamvaca,K.;Jelesarov,I.;Hilvert,D.J.Mol.Biol.,2008,382: 971 February 3,2010;Revised:April 1,2010;Published on Web:May 13,2010. Intrinsically Disordered Proteins:the New Sequence-Structure-Function Relations HUANG Yong-Qi LIU Zhi-Rong*(Beijing National Laboratory for Molecular Sciences,Center for Theoretical Biology,College of Chemistry and Molecular Engineering,Peking University,Beijing 100871,P.R.China) Intrinsically disordered proteins(IDPs)are a new class of proteins which lack a unique tertiary structure under native conditions while possessing essential biological functions.They take part in various physiological processes such as signal transduction,transcription and translation regulation,and protein modification.The discovery of IDPs challenges the conventional protein“sequence-structure-function”paradigm.In this review,we first overview the history of the conventional protein paradigm and the discovery of IDPs.Then we discuss the characteristics of IDPs in terms of sequence,structure,and biological function.Taking molecular recognition processes as an example,we further introduce current opinions on the advantages of IDPs in binding.Finally,we analyze possible applications of the study of IDPs such as further understanding the protein folding mechanism,improving protein structure determination,providing new clues for protein design and new targets for drug design.The current status of IDPs study in China is also briefly presented. Intrinsically disordered protein; Protein folding; Protein interaction; Molecular recognition; Protein structure prediction; Drug design [Review] www.whxb.pku.edu.cn *Corresponding author.Email:[email protected];Tel:+86-10-62752541;Fax:+86-10-62759595. The project was supported by the National Natural Science Foundation of China(20973016,10721403),National Key Basis Research Program of China(973)(2009CB918500),and Scientific Research Foundation for the Returned Overseas Chinese Scholars,Ministry of Education,China. 国家自然科学基金(20973016、10721403)、国家重点基础研究发展计划项目(973)(2009CB918500)和教育部留学回国人员科研启动基金资助 O641;Q51 猜你喜欢 构象无序蛋白质 车身无序堆叠零件自动抓取系统汽车实用技术(2022年14期)2022-07-30蛋白质自由肝博士(2022年3期)2022-06-30环境无序性对消费者多样化寻求的影响及作用机制*心理学报(2022年1期)2022-01-21人工智能与蛋白质结构海外星云(2021年9期)2021-10-14丝氨酸构象异构化机理的密度泛函理论研究唐山师范学院学报(2020年6期)2020-04-16张博庭:煤电不能再这么无序发展下去了能源(2017年11期)2017-12-13一种一枝黄花内酯分子结构与构象的计算研究温州大学学报(自然科学版)(2016年1期)2016-10-27无序体系中的国际秩序国际政治科学(2016年2期)2016-10-20玉米麸质阿拉伯木聚糖在水溶液中的聚集和构象应用化工(2014年7期)2014-08-09Cu2+/Mn2+存在下白花丹素对人血清白蛋白构象的影响无机化学学报(2014年5期)2014-02-28
|
【本文地址】




 物理化学学报2010年8期
物理化学学报2010年8期