| HMMER 官方文档学习笔记系列 | 您所在的位置:网站首页 › techtree编程 › HMMER 官方文档学习笔记系列 |
HMMER 官方文档学习笔记系列
|
HMMER 官方文档学习笔记系列
本文严禁在未征得作者同意的情况下进行转载 一、HMMER 简介及安装原文链接:HMMER 官方文档学习笔记系列——HMMER 简介及安装 1. 说明“MMER 官方文档学习笔记系列”是本人在阅读 HMMER 官方文档来学习 HMMER 使用方法的过程中整理的学习笔记类教程。该系列主要以 HMMER 官方文档为参考,因此包含很多对原文档的直接翻译和直接摘录。旨在通过转述官方文档说明,帮助需要使用该软件的人更快的上手使用。但本人并非生信专业出身,计算机相关知识也仅处于起步水平,业余时间也比较有限。因此在对原文档的理解、转译和论述会存在很多错误,在此欢迎有兴趣的小伙伴对次批评指正,一起探讨学习。官方文档下载地址:HMMER 官方文档。 2. 太长不看系列这一部分将对安装过程做简要概括,如果嫌弃本文章过长而又拥有比较扎实的 Linux 系统使用基础的话,可以选择仅看此处的安装概要;如果 Linux 系统相关使用技能比较薄弱的话,建议跳过该部分,从第 3 小节开始阅读全文。 2.1 在 Linux 或 Mac OS/X 系统上安装 HMMER 通过软件仓库安装$ sudo brew install hmmer # OS/X, HomeBrew $ sudo port install hmmer # OS/X, MacPorts $ sudo apt install hmmer # Linux (Ubuntu, Debian...) $ sudo dnf install hmmer # Linux (Fedora) $ sudo yum install hmmer # Linux (older Fedora) $ sudo conda install -c biocore hmmer # Anaconda 通过源码编译安装$ wget http://eddylab.org/software/hmmer/hmmer-3.3.2.tar.gz $ tar xf hmmer-3.3.2.tar.gz $ cd hmmer-3.3.2 $ ./configure --prefix=自定义目录 $ make $ make install $ cd easel #最后这两行代码为可选步骤,执行的话会安装一套被成为迷你应用(miniapps)的额外小工具 $ make install #最后这两行代码为可选步骤,执行的话会安装一套被成为迷你应用(miniapps)的额外小工具 安装后,修改 /etc/profile 文件,将软件添加到 PATH 环境变量中,以方便调用:$ sudo cp /etc/profile /etc/profile.bubackup $ sudo vim /etc/profile #在文档末尾加入: export PATH=自定义目录/bin:$PATH export MANPATH=自定义目录/share/man:$MANPATH 随后,重启电脑,或通过以下命令更新 PATH 环境变量:source /etc/profile 注意,该命令只会更新当前用户的 PATH 环境变量,如果切换用户,这需要重新执行该命令。如果重启了电脑,无需执行该命令,便会对所有用户生效。 2.1 在 Windows 系统上安装 HMMER 通过访问 http://eddylab.org/software/hmmer3/3.0/hmmer-3.0-windows.zip 链接进行下载软件压缩包(参考——HMMer在Windows环境下的安装_我就叫陌了这还能重名的博客-CSDN博客_hmmer安装)。解压缩后即可在解压目录中通过命令提示符界面(cmd)执行命令行运行。 3. HMMER 简介HMMER是一个软件包,提供了一类被称为轮廓隐马尔可夫模型(此处中文名为直译,英文原称为 profile hidden Markov models, profile HMMs 或 profiles)的工具,这些工具用于制作蛋白质和DNA序列域家族概率模型,以及可以使用这些工具来注释新的序列。不同于 Blast 等工具,HMMER 使用的是集成算法(ensemble algorithms),而不是最佳对齐 (optimal alignment)。进一步来说,HMMER 使用的是基于隐马尔可夫模型的一种算法,但具体的算法原理过于理论,本人目前难以深入理解,因此不过多论述。 轮廓隐马尔可夫模型的最初是通过计算密集型的算法来实现的,包括之前的 HMMER1(1995)和 HMMER2(1998)这两个版本,但最新的 HMMER3 版本通常比 BLASTP 或 FASTA 搜索的更快,即使它使用的模型比二者更复杂。HMMER 可用于进行灵敏的同源搜索、蛋白结构域的自动注释、深度多重比较数据集的管理等领域。此外 HMMER 3 不仅仅适用于多个比对,也适用于单序列比较。两两序列比较只是轮廓隐马尔可夫模型的一个特例。HMMER 可以使用 BLOSUM 替换矩阵来参数化仅从一个序列构建的序列谱隐马尔可夫模型。HMMER3 包含两个用于使用单个查询序列搜索蛋白质数据库的程序:phmmer 和 jackhmmer。研究人员相信 phmmer 在很多方面都优于 BLASTP,而 jackhmmer 在很多方面也优于 PSI-BLAST。 当然,任何方法都不可能是完美无缺的。HMMER 的算法没有考虑不同个体对象间的更高一级的相互作用,因此不适合应用于存在相互作用的模型分析(如:碱基和碱基之间的相互作用,会使一些 RNA 形成特殊结构,对于这些结构 RNA(此处为直译,英文原称:structural RNAs)的分析,HMMER 工具便不再适用了)。此外,HMMER 的算法也缺乏系统发育的相关模型,因此不适合用于进化树的构建。 4. HMMER 安装 4.1 在 Linux 系统上安装 HMMER 说明关于命令输入的一些说明:文中每条命令的前面都会带有 $ 或 #,这两个符号不是命令的组成部分,而是类似于身份标识(具体专业描述不知道是什么),比如我们打开终端,默认会有用户名@localhost:~$的前缀,而最后的 $ 符号便是提示使用者身份的标识。在下面所有的命令中,也采用 $ 或 # 进行身份标识,其中代码前带有 $ 符号的命令表示以普通用户身份执行命令代码;前标识为 # 的命令表示在超级管理员(root)身份下运行命令。而代码中其他地方带有的 # 多数情况下表示对命令的解释说明,也就是注释命令,同样也不属于命令的组成部分。 简言之,,每一条命令前的 $ 或 #,以及其他位置的 # 和这个 # 后面紧跟的同一行内容都是类似解释说明一类的东西,不属于命令本身,复制粘贴的话请忽略。 4.1.1 通过官方软件源进行安装 在 Ubuntu 系统中使用以下命令安装:$ sudo apt install hmmer 在 Debian 系统中的安装命令于此类似,但默认情况下,要先切换为管理员用户,再执行安装命令,而不能直接通过 sudo 命令安装:$ su - # apt install hmmer第一行 su - 命令即代表切换到超级管理员模式,并且切换后位于相应管理员用户的用户目录,即 /root 目录,如果要在当前目录切换使用超级管理员,需要输入 su root 进行切换。安装软件则通常使用 su - 命令切换。 在 Fedora 较新版本系统中,使用如下命令安装:$ sudo dnf install hmmer 较老版本的 Fedora 系统使用如下命令安装:$ sudo yum install hmmer由于本人目前仅在 openSUSE 系统上尝试安装成功,因此以上操作本人没有进行确认,仅将官方说明摘录于此。 4.1.2 通过第三方包管理库进行安装 通过 Anaconda 进行安装(首先确保系统已经安装了 Anaconda 包管理器):$ sudo conda install -c biocore hmmer由于本人目前仅在 openSUSE 系统上尝试安装成功,因此以上操作本人没有进行确认,仅将官方说明摘录于此。 4.1.3 通过源码编译进行安装HMMER 软件的源码编译安装十分简单,而且在多种 Linux 系统下通用,因此个人也比较推荐通过源码安装。而且通过源码安装,可以同时方便的同时安装 HMMER 2 版本和 HMMER 3 版本,这样就可以使用一些 HMMER 3 不具有的,而 HMMER 2 具有的功能。源码安装具体步骤如下: 下载源码安装压缩包 首先,通过 weget 命令行在 eddylab.org/software/hmmer/hmmer-3.3.2.tar.gz 路径获得,随后解压缩并通过 cd 命令进入解压后的 hmmer-3.3.2 目录: $ wget http://eddylab.org/software/hmmer/hmmer-3.3.2.tar.gz $ tar xf hmmer-3.3.2.tar.gz $ cd hmmer-3.3.2也可以去软件官网下载 .tar.gz 格式的安装包,然后解压缩并进入 解压目录。 安装前配置 然后,通过 ./configure 命令对 HMMER 的安装进行配置。命令格式如下: $ ./configure 参数这里的参数是可选项,也就是可以选择添加参数,也可以选择不添加。参数之间,以及参数和其他命令之间要有空格分开。参数的话主要有 3 类: 第一类类参数可以自定义安装目录。 如--prefix 参数可以自定义总的程序安装目录,如: $ ./configure --prefix=/your/install/path如上所示,如果自定义路径为 /your/install/path,那么程序和程序操作说明则会分别安装在 /your/install/path/bin 和 /your/install/path/share/man/man1/ 目录下。如果不设置该参数,程序和操作说明则会分别安装在默认的 /usr/local/bin 和 /usr/local/share/man/man1/ 目录。默认目录是按照 GNU 编码标准进行分布的,其他的目录也可以通过不同的参数修改,具体如下: 变量(Variable) 默认安装路径 ./configure 参数 prefix /usr/local --prefix exec_prefix prefix --exec_prefix bindir exec_prefix/bi --bindir datarootdir prefix/share --datarootdir mandir datarootdir/man --mandir man1dir mandir/man1 --man1dir 不过个人不建议进行复杂的目录修改,仅仅使用 --prefix 自定义安装目录就够了。第二类参数可以设置是否开启多线程模式。默认情况安装的软件是开启多线程的,如果想禁用多线程,可添加参数 –disable-threads ,开启多线程后,在安装后的软件使用中,线程数默认是 2,在每次使用时,可以通过添加参数 –cpu 或 HMMER_NCPU 环境变量来设置线程数 n。(这里我没有完全搞懂,不过像我一样似懂非懂或者不懂的话可以不使用该参数) 第三类参数可以控制 MPI(Message Passing Interface,消息传递接口)的开启或关闭。除了 nhmmer 和 nhmmscan 两个程序外,其他的程序都支持 MPI(Message Passing Interface,消息传递接口),但该服务默认是关闭的,如需启用,除了需要安装相应的 MPI 库外,还需要在执行 ./configure 命令时添加 –enable-mpi 参数。在使用 MPI 并行化( MPI parallelization)时,每次添加 –mpi 参数可以开启主/辅并行模式。hmmbuild 的 MPI 实现可以扩展到数百个处理器,hmmsearch 也可以进行类似扩展。其他搜索程序(hmmscan、phmmer 和 jackhmmer)的扩展性非常差,最多不应该在数十个处理器上使用。 进行编译 执行完上述 ./configure 命令后,等待终端执行完毕后,接着就可以执行 make 命令进行编译: $ make同样,make 命令也可以选择带参数运行,如:通过添加 check 参数,会在编译的时候同时运行检测程序: $ make checkmake 命令可以带的参数归纳如下: all:建造完整的程序。就像单独使用 make 命令一样; check: 在 HMMER 和 Easel 库中运行自动测试检查模块; pdf:编译该软件的用户指南; install:安装程序和相应的程序说明文档; uninstall:卸载 install 命令参数安装的程序和相应的程序说明文档; clean:删除编译(来自于 make 命令)生成的所有文件,但保留配置文件(由 ./configure 命令生成的文件); distclean:删除所有编译(来自于 make 命令)生成的所有文件,和配置文件(由 ./configure 命令生成的文件)。 默认情况下,make 使用从 git 源代码中"偷来"的漂亮包装来隐藏编译过程的实际情况。如果希望看到命令行在其编译时的真实丑陋的外观,请传递一个 V=1 参数(V表示“verbose”)来实现(来自官方文档作者的幽默自述): $ make V=1HMMER 的 make install 支持分阶段安装,支持打包程序用于指定 buildroot 的传统 DESTDIR 变量(这个没有用过,所以具体的意思也没有很明白,不过一般情况下,为了使用方便,还是推荐不使用此参数的完整安装): $ make DESTDIR=/rpm/tmp/buildroot install安装 HMMER 程序和程序说明文档 执行上述编译后, 便会在解压目录的 src 文件夹得到二进制程序文件,可以直接运行这些文件来运行程序,但是通常为了便于使用,会通过以下命令进行安装,以将应用程序和程序的说明文档安装在我们上面通过 ./configure 命令自定义的文件夹下: $ make install安装时如果选择了自定义路径,此时最好将自定义目录加入 PATH 环境变量。 如果通过 --prefix= 重新自定义了安装路径,为了以后在终端下的任何文件路径下都快可以直接调用,需要将自定义的 自定义目录/bin 添加到 PATH 路径中,将 自定义目录/share/man 添加到 MANPATH 路径中,以方便在命令行使用软件和调出帮助文档。比如上面的例子我们的自定义目录为/your/install/path,则可以在终端执行下面的命令来完成该操作: $ export PATH=/your/install/path/bin:$PATH $ export MANPATH=/your/install/path/share/man:$MANPATH但是用种方法添加之后,仅仅对当前用户生效,而且重启电脑后需要重新配置。若想对全部用户生效,需要修改 /etc/profile 文件(下面第一行是复制命令,以实现在操作前对该文件进行备份;vim 是一种比较方便的命令行文本编辑器,虽然好用,但是和我们平常的文本编辑器的使用有差异,在执行下面命令之前,最好确保自己已经会一些 vim 编辑器的基本操作): $ sudo cp /etc/profile /etc/profile.bubackup $ sudo vim /etc/profile #在文档末尾加入: export PATH=/your/install/path/bin:$PATH export MANPATH=/your/install/path/share/man:$MANPATH随后,重启电脑,或通过以下命令更新 PATH 环境变量: source /etc/profile注意,该命令只会更新当前用户的 PATH 环境变量,如果切换用户,这需要重新执行该命令。如果重启了电脑,无需执行该命令,便会对所有用户生效。 如果不进行此设置,每次使用需要使用 cd 命令切换到自定义文件夹下的 bin 目录调用相关程序。到这里,HMMER 程序就基本安装好了。 可选步骤:安装额外的小工具 另外,还可以选择安装一套被成为迷你应用(miniapps)的额外小工具,安装时先通过 cd 进入解压目录下的 easel 文件夹,然后执行 make install 命令安装 miniapps 及其使用说明: $ cd easel $ make install源码编译常见错误 如果自定义目录的编译执行失败,有可能是 make 版本不支持自定义目录的构建,可尝试默认目录安装,或尝试安装 GNU make; 配置失败,提示“找不到可接受的 grep”。很多人已经在 Sun Sparc/Solaris 机器上发现了这种情况。这是 GNU autoconf 中的一个已知问题。可以通过安装 GNU grep ,或者坚持执行 ./configure 命令时将 GREP 设置为可工作的路径:$ ./configure GREP=/usr/xpg4/bin/grep 许多“检查”测试失败。如果不能正确的连接多线程,而引发测试错误,则可以试着关闭多线程:$ ./configure --disable-threads常见错误这一部分,由于本人安装过程比较顺利,没有遇到什么问题,因此也只是将官方文档提到的常见错误摘录出来。 同时安装 HMMER2 和 HMMER3 虽然 HMMER3 绝大部分功能都优于 HMMER2,但是 HMMER2 是可以进行全局和全局比对(global and glocal alignment),而 HMMER3 仅支持局部比对(local alignment),有时候,全局比对也是我们需要的,因此安装两个版本可以拓展适用范围(这里提到的全局比对和局部比对涉及到算法问题,本人水平还没涉及此高度,因此也没有完全理解,为了避免个人翻译错误,将原英文名称标注在括号里)。作者表示,在未来的 HMMER4 中,将同时支持两种比对方式。但是 HMMER2 和 HMMER3 由于存在相同的程序名,因此直接安装会出现程序名冲突,为了解决冲突,作者给了两个建议方案: 方案一,将两个版本的软件分别装在两个自定义路径,一个版本加入 PATH 环境变量,通过命令行直接使用;另一个版本则进入相应安装路径使用; 方案二,安装两个版本软件,将一个版本加入 PATH 环境变量后,再将另一个版本的相应文件复制并重命名到该版本路径。比如,假设我们自定义安装 HMMER 2 的目录为 /home/my/install/path/hmmer2,自定义安装 HMMER 3 的目录为 /home/my/install/path/HMMER3,则可以先将 HMMER 3 的相应目录按照上面的方法加入 PATH 环境变量,然后通过 cd 命令先进入 HMMER 2 的安装目录,然后通过 cp 命令复制并重命名到 HMMER 3 的安装目录:$ cd /home/my/install/path/hmmer2/bin $ cp hmmalign /home/my/install/path/HMMER3/bin/h2-hmmalign $ cp hmmconvert /home/my/install/path/HMMER3/bin/h2-hmmconvert $ cp …… …… 但是一个一个的复制比较麻烦,所以可以优化一下方案:首先通过 cd 先进入 HMMER 2 的安装目录,然后批量重命名,接着移动文件到 HMMER 3 目录。这时重命名和移动命令可以如下(批量重命名的命令在第二行代码和第五行代码,这两行代码都是在当前目录下的所有文件名之前添加“v2-”的前缀,另外注意这两行代码中的单引号不是普通单引号,而是反引号):$ cd /home/ligareen/Documents/software/hmmer/hmmer2/bin/ $ for i in `ls`; do mv -f $i `echo "v2-"$i`; done $ mv * /home/ligareen/Documents/software/hmmer/bin/ $ cd /home/ligareen/Documents/software/hmmer/hummer2/man/man1/ $ for i in `ls`; do mv -f $i `echo "v2-"$i`; done $ mv * /home/ligareen/Documents/software/hmmer/share/man/man1/此处的批量重命名操作参考:Linux 批量修改文件名(前缀或后缀)_@羲凡—只为更好的活着-CSDN博客_linux批量修改文件名前缀。 安装后简单调试 解压目录下的 tutorial 文件夹下,是一些示例文件,在其中,有一个 Stockholm 格式(后缀为 .sto)的序列比对文件,可以通过 hmmbuild 命令从这一份序列比对文件中构建一个轮廓隐马尔可夫模型(profile): $ cd tutorial $ hmmbuild globins4.hmm globins4.sto上述命令时在已经进入了解压目录后进行的操作。 正常的话会有以下输出: 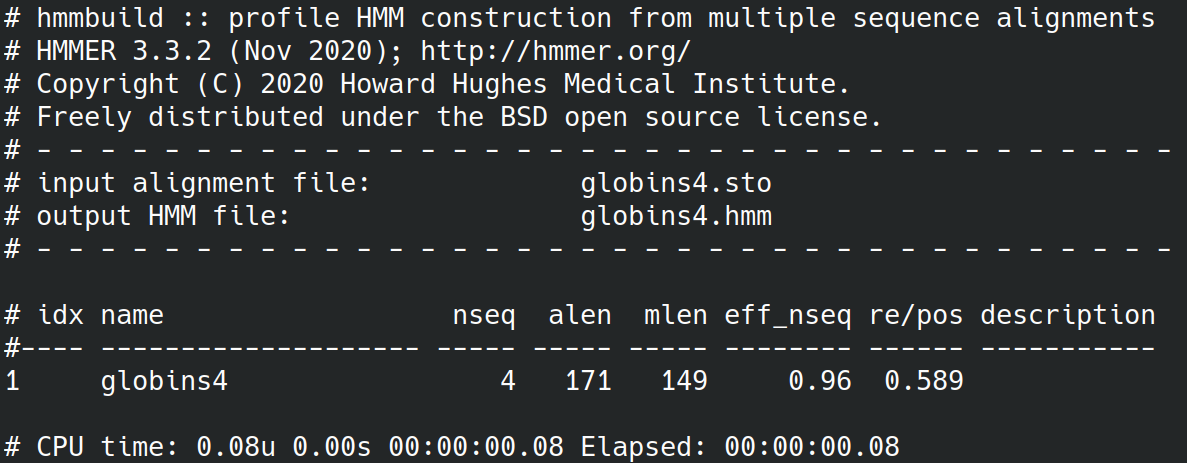
hmmsearch 命令可以针对数据库,基于已有的轮廓隐马尔可夫模型库进行轮廓隐马尔可夫模型的搜索。在解压目录下的 tutorial/globins45.fa 文件是包含45个珠蛋白序列的一个小型 FASTA 文件,利用上一条产生的轮廓隐马尔可夫模型(profile),可以通过以下命令对该文件进行序列谱隐马尔可夫模型的搜索: $ hmmsearch globins4.hmm globins45.fa这将直接显示一个搜索结果的输出,以表格的形式显示它们的对齐信息(输出结果过长。下图仅截取开头部分): 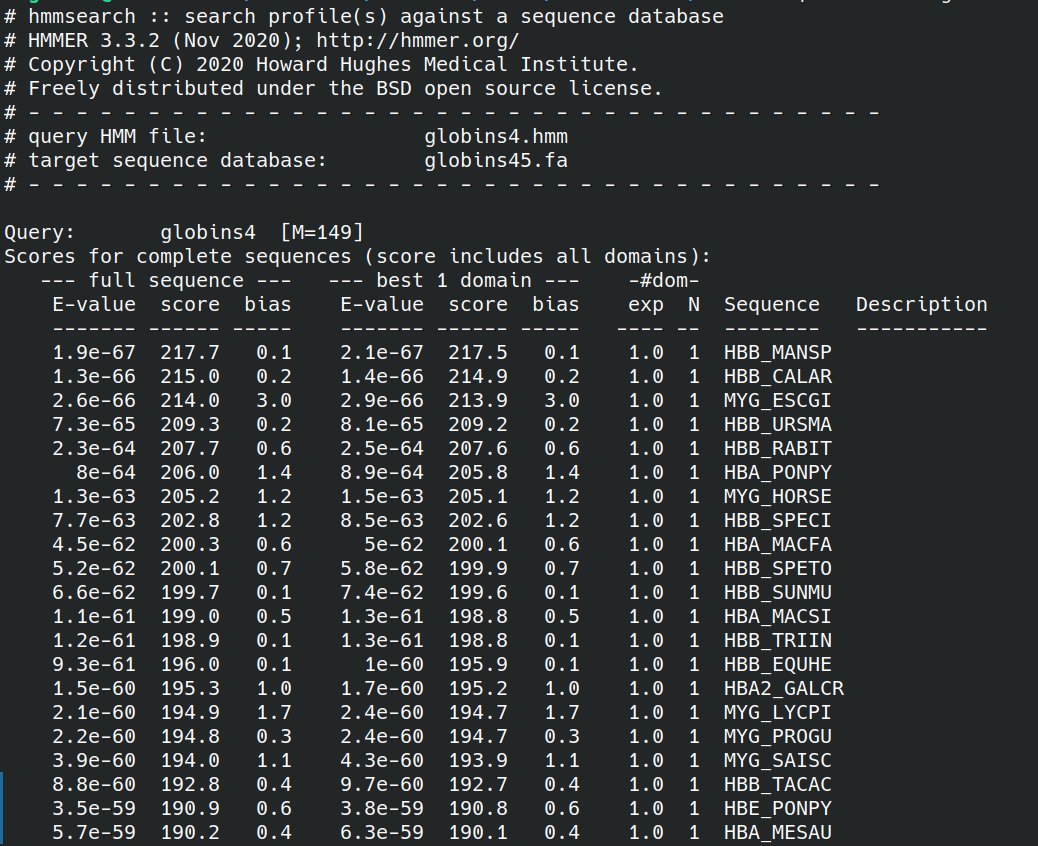 4.2 在 Mac OS/X 系统上安装 HMMER
4.2 在 Mac OS/X 系统上安装 HMMER
说明:由于本人没有使用过 Mac 系统,因此仅将官方文档中描述的安装方法摘录展示于此。 4.2.1 通过软件仓库进行安装 通过 HomeBrew 进行安装$ brew install hmmer 通过 MacPorts 进行安装$ port install hmmer 4.2.2 通过源码编译进行安装 源码编译安装的过程参考 Linux 系统下通过源码编译进行安装的过程(章节 1.3)。 4.3. 在 Windows 系统上安装 HMMER 4.3.1 下载安装包后解压直接使用首先去官网下载 Windows 版本的压缩包。由于目前官网的最新版本不支持 Windows 系统,所以官网并没有直接的下载链接,在这里,参考——HMMer在Windows环境下的安装_我就叫陌了这还能重名的博客-CSDN博客_hmmer安装(不得不感慨,这位网友真的太有才了!) ,通过访问 http://eddylab.org/software/hmmer3/3.0/hmmer-3.0-windows.zip 链接进行压缩包的下载。 下载好后解压到任意目录,便可以直接使用,但双击程序多半会闪退。其实在这里,使用方法和 Linux 下的方法是一样的,需要在命令提示符(cmd,类似 Linux 下的终端)下通过命令行进行使用。(Windows 系统下调出 cmd 的过程可以是:按快捷键 win+R 快捷键调出“运行”窗口后输入“cmd”后按回车键。) 在 cmd 下使用时,类似 Linux 系统,需要通过 cd 命令进入解压文件夹使用,如通过 hmmpress,exe -h 命令查看 hmmpress,exe 程序的帮助信息(下列代码中,“>”号不是命令的组成部分): > cd C:\Users\username\Downloads\hmmer-3.0-windows > hmmpress,exe -h这样每次都要进入解压目录才能调用,会让人感觉很麻烦,在 Windows 下也可以像 Linux 系统一样,将程序添加到 PATH 环境,添加后可以在任意目录下调用,添加步骤如下(以 win11 为例,win10 适用,大部分图片点击可看大图,步骤参考【图文详解】环境变量中Path环境变量作用详解_二十四日的博客-CSDN博客): 通过 系统设置→系统→关于→高级系统设置 步骤进入高级设置:

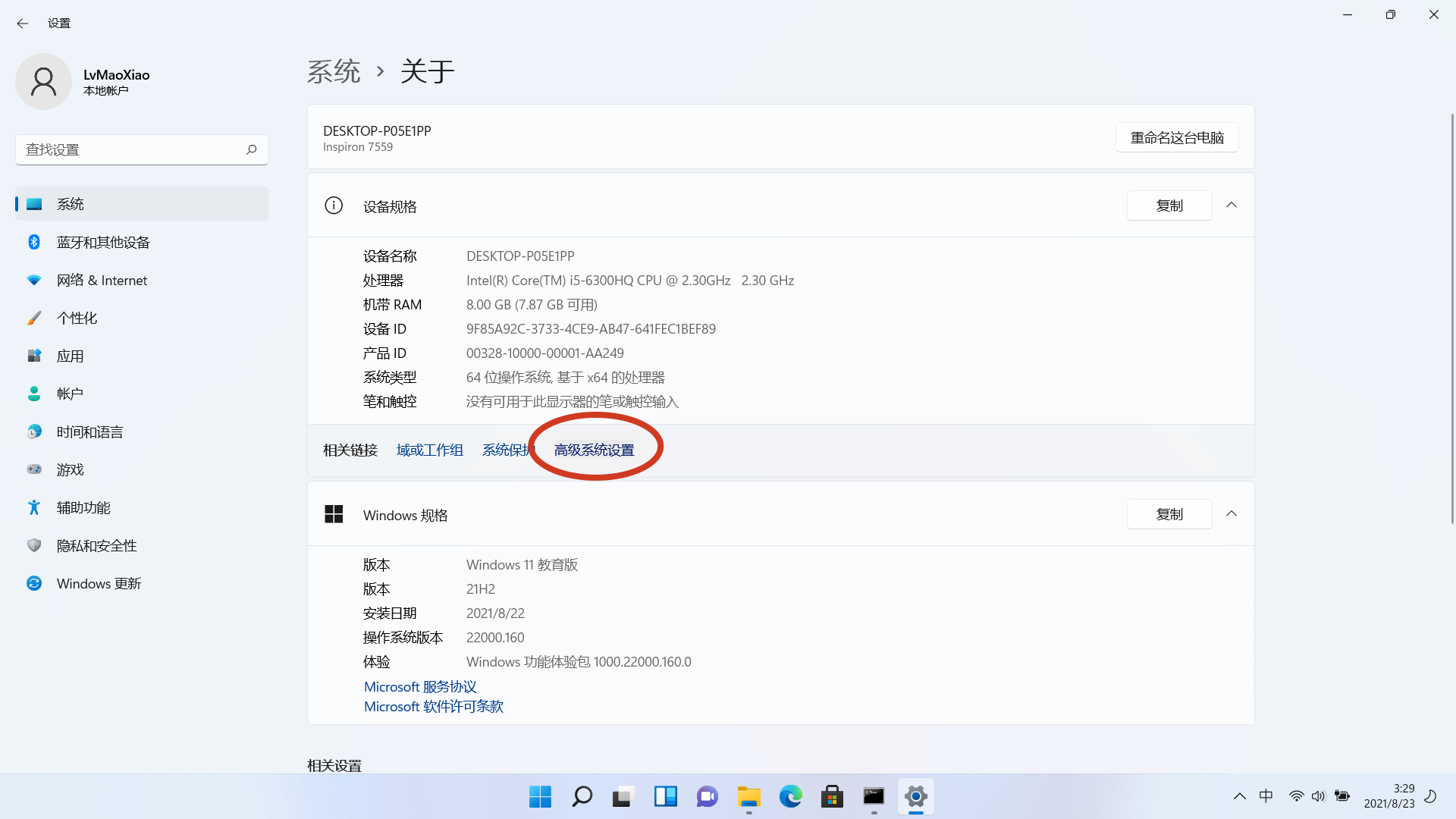 在弹出的程序框中选择环境变量选项:
在弹出的程序框中选择环境变量选项:
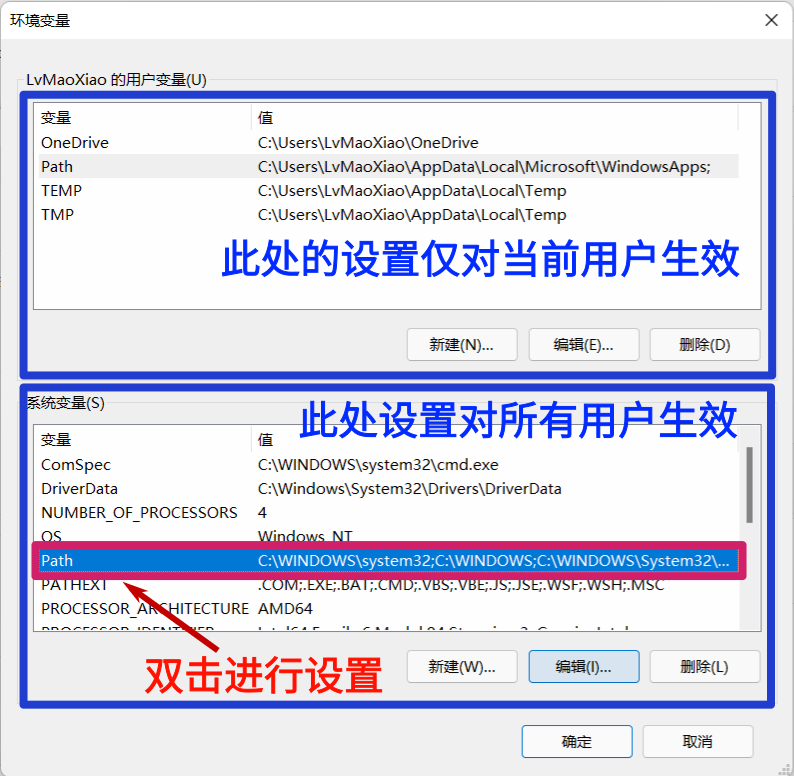
 随后新弹出的程序框便是环境变量设置对话框了。该对话框整体分为上下两部分,上面的那部分设置仅对当前用户生效,下面的设置对所有用户生效。因为我们一般的个人电脑仅会设置一个账户,不存在不同用户适用性的考量,但是,为了当便起见,如不涉及隐私问题,我们一般设置对全部用户生效。所以我们选择下面那一部分进行设置。找到并双击 Path 选项(或单击选择 Path 选项后,再单击编辑按钮):
随后新弹出的程序框便是环境变量设置对话框了。该对话框整体分为上下两部分,上面的那部分设置仅对当前用户生效,下面的设置对所有用户生效。因为我们一般的个人电脑仅会设置一个账户,不存在不同用户适用性的考量,但是,为了当便起见,如不涉及隐私问题,我们一般设置对全部用户生效。所以我们选择下面那一部分进行设置。找到并双击 Path 选项(或单击选择 Path 选项后,再单击编辑按钮):
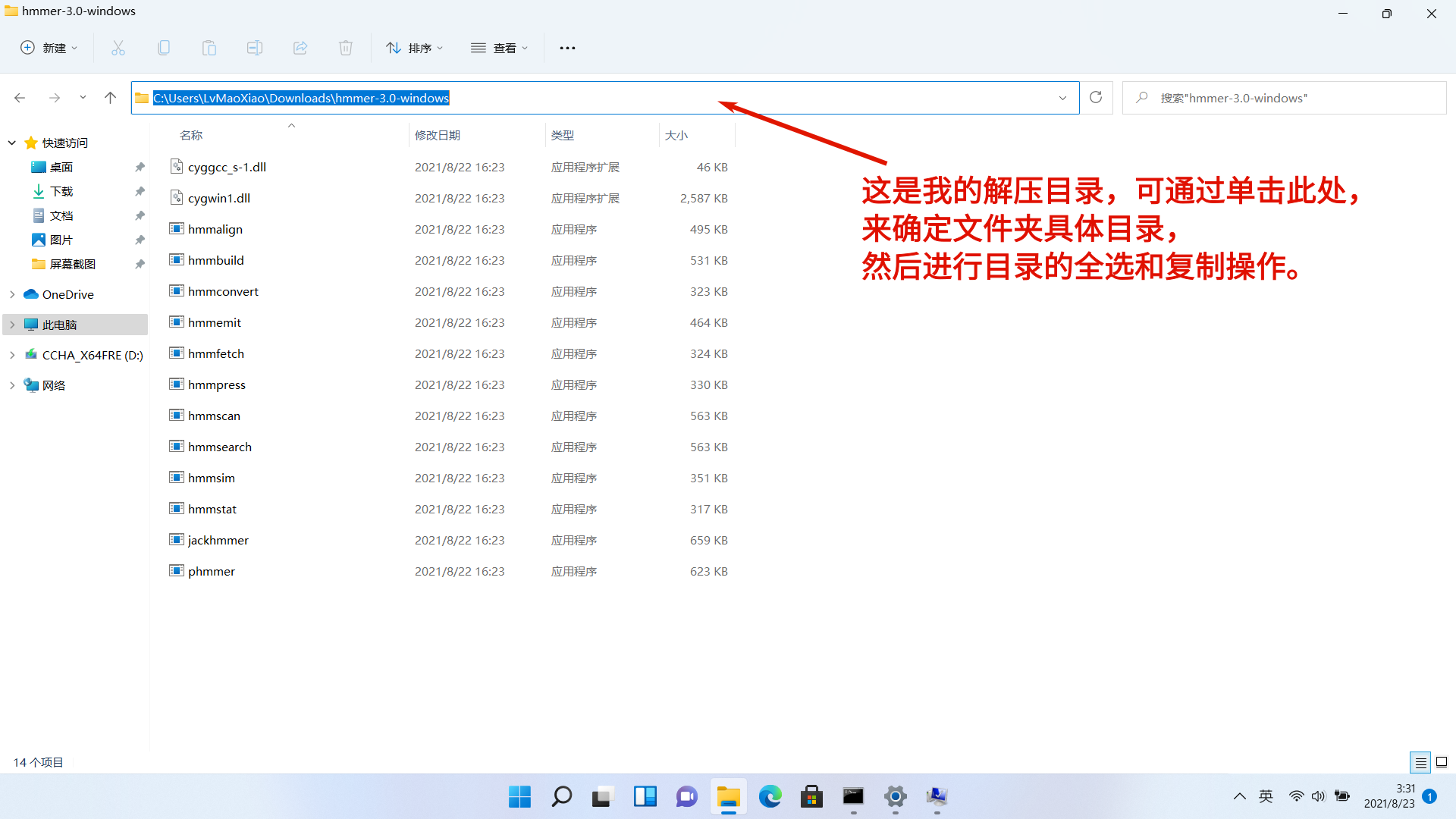
 然后在文件浏览器中查看并复制 HMMER 程序所在目录(即压缩包解压文件所在目录),然后。在上一步操作后新弹出的窗口中,通过新增 选项将刚刚复制的目录添加到 Path 环境,并单击确定按钮进行保存:
然后在文件浏览器中查看并复制 HMMER 程序所在目录(即压缩包解压文件所在目录),然后。在上一步操作后新弹出的窗口中,通过新增 选项将刚刚复制的目录添加到 Path 环境,并单击确定按钮进行保存:
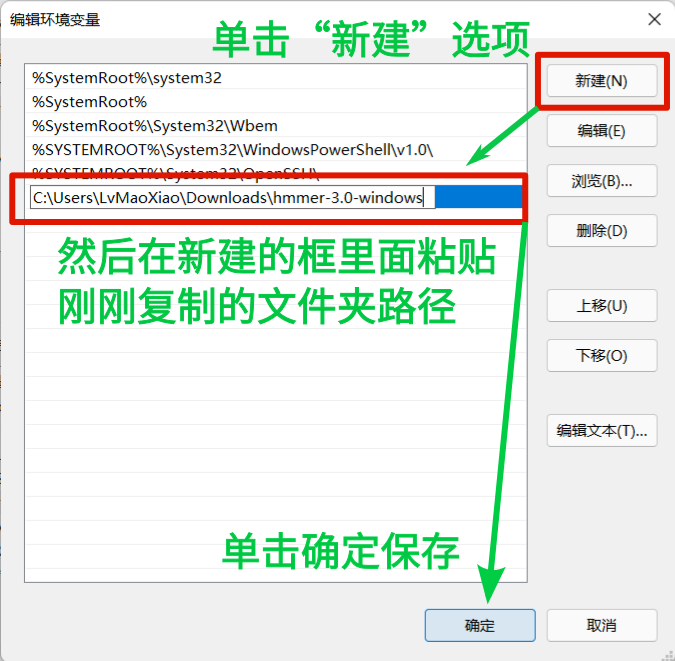 上述步骤进行完后,便已经将环境变量设置好了。下面进行简单的测试:
通过 Win+R 快捷键调出“运行”窗口后,输入 cmd 后回车,调出 DOS(命令提示符)界面:
上述步骤进行完后,便已经将环境变量设置好了。下面进行简单的测试:
通过 Win+R 快捷键调出“运行”窗口后,输入 cmd 后回车,调出 DOS(命令提示符)界面:
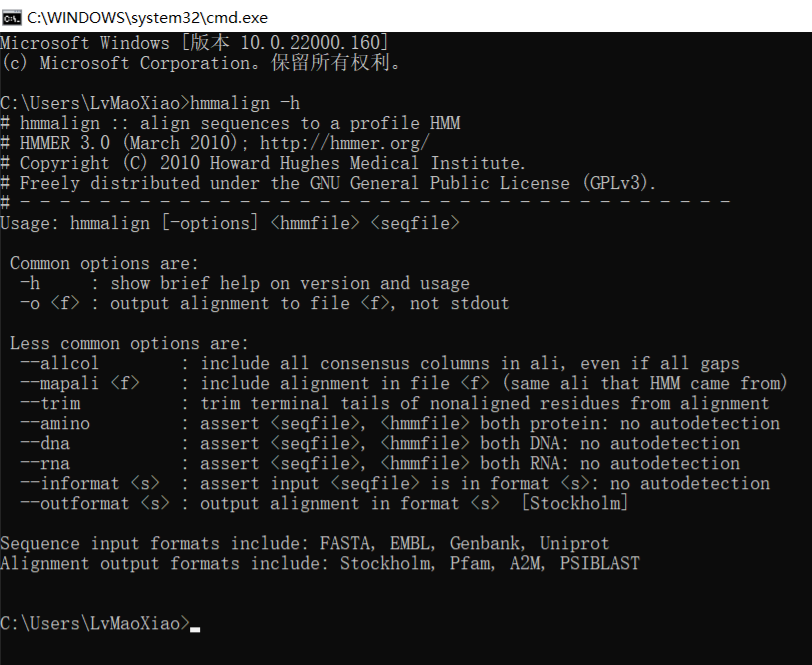
 在 DOS 中,不切换文件夹,直接执行 hmmalign -h 来查询帮助说明,执行后,如果一切正常,会有帮助文档的输出信息。
在 DOS 中,不切换文件夹,直接执行 hmmalign -h 来查询帮助说明,执行后,如果一切正常,会有帮助文档的输出信息。
 将 HMMER 添加到环境变量后,使用时不但不需要通过cd 命令进入相应文件夹再使用,而且在使用的过程中,程序名省略了后缀 .exe,使用方便了很多。但是程序名却不能通过 TAB 键补全,只能全拼写入(正常情况下,不管时 windows 系统还是 Linux 系统,我们在输入命令时,往往可以在输入开头几个字母后,按 TAB 键补全后续的文件名)。
将 HMMER 添加到环境变量后,使用时不但不需要通过cd 命令进入相应文件夹再使用,而且在使用的过程中,程序名省略了后缀 .exe,使用方便了很多。但是程序名却不能通过 TAB 键补全,只能全拼写入(正常情况下,不管时 windows 系统还是 Linux 系统,我们在输入命令时,往往可以在输入开头几个字母后,按 TAB 键补全后续的文件名)。
很多生信软件在 Linux 系统上安装和使用都很方便,就像 HMMER,通过 Linux 系统,不但可以安装最新版本,获得更完善的程序功能,而且可以方便的将软件加入 PATH 变量环境,并可以方便调用(如通过 TAB 键快速补全程序名)。因此,总体而言,个人推荐通过 Linux 系统使用该软件。 结束第一期的 HMMER 简介和安装到此就结束了,后面几期会介绍 HMMER 的使用方法。希望大家都能从我的文章中有所收获~ 参考[1] HMMER 官网 [2] HMMER 官方文档 [3] linux增加自定义path和manpath_flora_yao的博客-CSDN博客 [4] inux 批量修改文件名(前缀或后缀)_@羲凡—只为更好的活着-CSDN博客_linux批量修改文件名前缀 [5] HMMer在Windows环境下的安装_我就叫陌了这还能重名的博客-CSDN博客_hmmer安装 [6] 【图文详解】环境变量中Path环境变量作用详解_二十四日的博客-CSDN博客 [7] Linux将命令添加到PATH中 - 天道酬勤 - 博客园 HMMER 官方文档学习笔记系列 一、HMMER 简介及安装 二、HMMER 软件包使用概述(预计2021年10月20日前成稿) 二、通过隐马尔可夫模型文件搜索序列数据库(预计2021年10月31日前成稿)
|
【本文地址】